Original:
Coronavirus Pandemic—Therapy and
Vaccines
PanTherapeutics,
CH1095 Lutry, Switzerland
Received: 31 March 2020 /
Revised: 29 April 2020 / Accepted: 30 April 2020 /
Published: 3 May 2020
The current coronavirus
COVID-19 pandemic, which originated in Wuhan,
China, has raised significant social,
psychological and economic concerns in addition to
direct medical issues. The rapid spread of severe
acute respiratory syndrome-coronavirus
(SARS-CoV)-2 to almost every country on the globe
and the failure to contain the infections have
contributed to fear and panic worldwide. The lack
of available and efficient antiviral drugs or
vaccines has further worsened the situation. For
these reasons, it cannot be overstated that an
accelerated effort for the development of novel
drugs and vaccines is needed. In this context,
novel approaches in both gene therapy and vaccine
development are essential. Previous experience
from SARS- and MERS-coronavirus vaccine and drug
development projects have targeted glycoprotein
epitopes, monoclonal antibodies, angiotensin
receptor blockers and gene silencing technologies,
which may be useful for COVID-19 too. Moreover,
existing antivirals used for other types of viral
infections have been considered as urgent action
is necessary. This review aims at providing a
background of coronavirus genetics and biology,
examples of therapeutic and vaccine strategies
taken and potential innovative novel approaches in
progress.
1.
Introduction [coronavirus also with animals]
Coronaviruses belong to
the family Coronaviridae
and are characterized by causing respiratory
tract infections ranging from mild diseases
such as common cold to pneumonia with a
lethal outcome [ 1].
Typically, coronaviruses have been
associated with a large number of diseases
in livestock and companion animals such as
pigs, cows, chickens, cats and dogs ()
[ 2]. In this
context, transmissible gastroenteritis virus
(TGEV) [ 3] and
porcine epidemic diarrhea virus (PEDV) [ 4] are
responsible for significant morbidity and
mortality in young piglets. Similarly,
porcine hemagglutinating encephalomyelitis
virus (PHEV) causes enteric infection in
pigs but can also lead to encephalitis by
targeting the nervous system [ 5]. In
cattle, bovine CoV (BCoV) is responsible for
mild to severe respiratory tract infections,
resulting in significant losses in the
cattle industry due to diarrhea,
dehydration, decreased milk production and
depression [ 6, 7]. In
addition to cattle, BCoV also infects other
ruminants such as elk, deer and camels.
Another coronavirus, rat CoV (RCoV), causes
respiratory tract infections in rats,
providing a useful model for studying early
events of innate immune responses to
coronavirus infections in lungs [ 8].
Infectious bronchitis virus (IBV) targets
chickens, causing respiratory tract
infections but also renal disease [ 9]. IBV has
a significant negative effect on egg
production and growth of chickens, leading
to substantial losses in the chicken
industry [ 7]. In
domestic cats, a mild or asymptomatic
infection has been associated with feline
enteric coronavirus (FCoV) [ 10],
although a highly virulent strain of feline
infectious peritonitis virus (FIPV) causes
lethal feline infectious peritonitis (FIP) [ 11], which
shows similarities to human sarcoidosis [ 12].
Table
1. Coronavirus-based diseases in
animals and humans.
Interestingly, wildlife
can also be infected by coronaviruses. For
instance, a novel coronavirus SW1 was
isolated from a deceased Beluga whale [ 13].
The whale had suffered from a respiratory
disease and acute liver failure and large
quantities of SW1 particles were isolated
from the liver. However, electron microscopy
was not able to identify the virus as a
coronavirus, but nucleic acid sequencing of
liver tissue confirmed the presence of
coronavirus RNA. Phylogenetic analysis
indicated that SW1 belonged to the group of
γ-coronaviruses.
[Coronavirus19 and bats]
For obvious reasons, there has been an
intense interest in bat coronaviruses, as
bats have been indicated as the original
source of several coronavirus outbreaks. In
the past decade, hundreds of novel bat
coronaviruses have been identified [ 14]. In
this context, both the severe acute
respiratory syndrome-coronavirus (SARS-CoV)
and the Middle East respiratory syndrome-CoV
(MERS-CoV) have been suggested to originate
from bats. Moreover, it has been already
discussed in 2015 that a SARS-like cluster
of bat coronaviruses could pose a threat for
human epidemics [ 15]. For
instance, the disease potential of a
SARS-like virus, SHC014-CoV, circulating in
Chinese horseshoe bats was evaluated by
reverse genetics [ 15]. The
chimeric virus composed of SHC014-CoV spikes
and wild-type backbone was able to
efficiently utilize multiple orthologs of
the SARS receptor, human
angiotensin-converting enzyme 2 (ACE2),
replicate to high levels in primary human
airway cells and generate in vitro titers
equivalent to epidemic SARS-CoV strains.
Moreover, the chimeric virus showed
replication in mouse lung, with notable
pathogenesis in vivo. These results
indicated that there is a potential risk of
SARS-CoV re-emergence from viruses
circulating in bat populations. Needless to
say, the re-emergence of SARS-CoV is a
reality today, as will be discussed below.
The murine hepatitis virus
(MHV) is the best studied animal coronavirus
resulting in respiratory, enteric, hepatic
and neurological infections and is also a
useful disease model [ 16]. For
instance, MHV-1 causes severe respiratory
infections in A/J and C3H/HeI mice. A59 and
MHV-3 are associated with severe hepatitis,
and JHMV causes severe encephalitis.
Interestingly, A59 and an attenuated JHMV
cause chronic demyelinating disease in mice,
which resembles multiple sclerosis (MS),
providing a mouse model for MS [ 17].
Another advantage of MHV is the requirement
of BSL2-level laboratory conditions, whereas
SARS-CoV and MERS-CoV require BSL3-level
laboratory conditions, making MHV ideal for
studies on replication in cell cultures as
well as pathogenesis and immune responses in
vivo.
In the context of human
coronaviruses, it was thought that they
caused only mild self-limiting infections
until the SARS-CoV outbreak in 2002–2003 [ 2]. Two
human α-coronaviruses (HCoV-229E and
HCoV-NL63) and two β-coronaviruses
(HCoV-OC43 and HCoV-HKU1) were identified as
endemic in human populations, responsible
for 15%–30% of annual respiratory tract
infections [ 18, 20].
However, a more severe disease has been
detected in neonates, elderly people and in
individuals with pre-existing illnesses.
Moreover, HCoV-NL63 has also been associated
with acute laryngotracheitis [ 19].
Interestingly, human coronaviruses differ in
genetic variability, as HCoV-229E isolated
from around the world showed only minimal
sequence divergence [ 23, 24, 25, 26, 27, 28, 29, 30], while
HCoV-OC43 isolated from the same location in
different years demonstrated significant
genetic variability [ 31]. For
this reason, HCoV-229E does not cross
species barriers and is incapable of
infecting mice, while mice and several
ruminant species are susceptible to
HCoV-OC43 and the closely related BCoV. As
mentioned above, although MHV possesses the
ability to cause demyelinating disease, no
evidence exists of human coronaviruses being
associated with MS.
[Coronavirus SARS 2002-2003]
The first major human
coronavirus outbreak caused by the
β-coronavirus SARS-CoV originated in
Guangdong in China [ 21]. During
the 2002–2003 epidemic, approximately 8098
cases were recorded, with 774 deaths at a
mortality rate of 9% [ 22].
However, the mortality rate was much higher
in the elderly population, reaching almost
50% in individuals over 60 years of age.
Although closely related viruses were
isolated from exotic animals such as
Himalayan palm civets and raccoon dogs [ 24], based
on sequence data and serologic evidence,
SARS-CoV originated in Chinese horseshoe
bats [ 25, 26].
Related to SARS-CoV, two novel bat
SARS-related CoVs with closer similarity to
SARS-CoV than any other virus, were
identified in 2013 [ 27]. They
utilize the same receptor, ACE2, as
SARS-CoV, another indication that SARS-CoV
originated in bats. The SARS outbreak was
only spread through direct contact with
infected individuals due to the relatively
inefficient transmission of the virus, which
allowed containing the outbreak within
households and health care institutions [ 28].
Therefore, the SARS-CoV outbreak was
controllable through quarantining, and it
died out in June 2003.
After the SARS-CoV
epidemic, the novel human MERS-CoV emerged
in the Middle East in 2012, causing a series
of highly pathogenic respiratory tract
infections in Saudi Arabia and other Middle
East countries [ 29].
Despite fears, the outbreak did not
accelerate in 2013 and the cases of MERS-CoV
totaled at 855 individuals and 333 deaths,
resulting in a mortality rate of almost 40%
[ 23].
MERS-CoV is a β-coronavirus, which is highly
related to the bat coronaviruses HKU4 and
HKU5 [ 32],
suggesting its origin in bats, although
dromedary camels have been indicated as an
intermediate host [ 33].
Moreover, it was demonstrated that MERS-CoV
replicated in camel cell lines, further
indicating that camels may be natural hosts
for the virus [ 34].
Additionally, a case study showed that virus
isolated from a person who had been in
contact with an infected camel was identical
to virus isolated from the camel [ 35]. In
contrast to SARS-CoV, the dipeptidyl
peptidase 4 (DPP4) receptor is utilized,
making bats, humans, camels, rabbits, and
horses susceptible [ 36].
However, differences in the structure of the
mouse DPP4 receptor make mouse cells
resistant to MERS-CoV, which does not allow
evaluation of antivirals and vaccines in
appropriate mouse models. However, a mouse
model for MERS-CoV was engineered by
introduction of the human DPP4 gene into
mouse lungs by an adenoviral vector [ 37].
The current 2019-nCoV,
officially called severe acute respiratory
virus-coronavirus-2 (SARS-CoV-2) was first
detected in the city of Wuhan in China in
December 2019 [ 38, 39]. It was
thought to have originated from wild animals
at the Huanan market in Wuhan and spread by
person-to-person transmission, causing the
disease named COVID-19, with various degrees
of severity, from mild flu-like symptoms to
pneumonia and death [ 39]. Bats,
snakes and pangolins are potential carriers
of SARS-CoV-2 based on sequence comparison
to virus isolated from SARS-CoV-2-infected
patients. The COVID-19 pandemic has already
overtaken both SARS and MERS in severity and
has recently led to extreme quarantine
measures including sealing off large cities,
closing borders and confining people to
their homes. Despite these efforts, as of
April 28, there are 3,041,912 cases of
COVID-19 and there have been 211,167 deaths
in 210 countries [ 40].
Whether the trend will be a further
expansion of the pandemic and when it will
die out or return in a seasonal pattern is
impossible to determine at this stage and
only time will tell the outcome.
Currently, there are no
antiviral drugs or vaccines available for
the treatment of COVID-19 [ 41]. The
urgent need for treatment has therefore
triggered a variety of approaches to design
and develop novel drug and vaccine
strategies against COVID-19. Therapeutic
approaches have included angiotensin
receptor blockers, monoclonal antibodies
(mAbs), gene silencing and even plant and
mushroom extracts based on traditional
Chinese medicine. Moreover, vaccine
development involves different alternatives
including epitope-based peptide vaccines,
viral vector-based vaccines and the support
of bioinformatics and immunoinformatics for
the design of more efficient targets.
However, before the various approaches are
described in detail, it is appropriate to
give an overview of the genomic composition
of SARS-CoV-2 and its lifecycle to identify
potential drug and vaccine targets. 2.
Coronavirus Genome and Lifecycle
Coronaviruses possess a
non-segmented positive-sense single-stranded
RNA (ssRNA) genome of approximately 30 kb [ 42, 43]. It
contains a 5′ end cap structure, replicase
genes (rep 1a and rep 1b) coding for the
non-structural proteins (nsPs) comprising
two-thirds of the genome and the structural
protein genes S (spike), E (envelope), M
(membrane) and N (nucleocapsid) and various
accessory genes interspersed within the
structural genes at the 3′ end [ 2] ().
Although the accessory proteins have been
considered non-essential for replication,
they play an important role in viral
pathogenesis [ 43].
Figure
1. Schematic illustration of the
severe acute respiratory
syndrome-coronavirus (SARS-CoV)-2 genome.
ORF1a and ORF1b encode the non-structural
proteins. The structural proteins are
encoded by spike (S), envelope (E),
membrane (M) and nucleocapsid (N) genes.
The virion structure has
been studied by cryo-electron tomography and
microscopy, showing the prominent
club-shaped spike projections on spherical
particles of approximately 125 nm in
diameter [ 44, 45]. The N
protein is helically symmetrical, with both
N- and C-terminal domains needed for optimal
encapsulation of the RNA genome [ 46]. The
transcription regulation sequences (TRSs) [ 47] and the
genomic packaging signal [ 48] have
been identified—of which, the latter has
been demonstrated to bind specifically to
the C-terminal RNA-binding domain
facilitating the packaging of the genome
into viral particles [ 49]. The M
protein containing three transmembrane
domains may exist as a dimer, binds to the N
protein [ 50] and is
thought to give the virion its shape [ 51]. The
topology of the small 8–12 kD E protein is
not completely resolved but is believed to
have a transmembrane structure with an
N-terminal ectodomain and a C-terminal
endodomain with ion channel activity [ 52]. It
facilitates the assembly and release of
virions and has been associated with
pathogenesis [ 53]. The
spikes consisting of the S protein give the
virion the appearance of a solar corona and
mediate the attachment to the host receptor
[ 54]. The S
protein is cleaved by a host cell furin-like
protease into S1 and S2 polypeptides for
most coronaviruses [ 55, 56].
However, SARS-CoV and SARS-related CoV lack
the furin cleavage site, while SARS-CoV-2
accommodates it [ 57]. A
fifth structural protein, the
hemagglutinin-esterase (HE), is present in
the genome of some coronaviruses such as MHV
[ 58]. The
function of the HE is thought to be the
enhancement of the S protein-mediated cell
entry and spread through the mucosa [ 59] as well
as increase of the neurovirulence of MHV [ 60].
The coronavirus lifecycle
is characterized by various steps including
attachment and entry, expression of
replicase proteins, replication and
transcription, and assembly and release of
mature viral particles ().
The initial attachment occurs between the
receptor-binding domain (RBD) of the S1
region of the S protein and its receptor.
MHV uses an RBD at the N-terminus [ 61], while
SARS-CoV has an RBD at the C-terminus of the
S1 region [ 62]. The S
protein–receptor interaction governs the
virus tropism, which also reflects the
targeting of different receptors by
different coronaviruses. For instance, MHV
targets carcinoembryonic antigen-related
adhesion molecule 1 (CEACAM1) [ 63],
MERS-CoV dipeptidyl-peptidase 4 (DPP4) [ 64] and
SARS-CoV and HCoVNL63 ACE2 [ 65].
Recently, it was demonstrated that
SARS-CoV-2 also uses ACE2 [ 21]. After
the initial attachment, acid-dependent
proteolytic cleavage of the S protein by
cathepsin or another protease occurs,
followed by fusion in acidified endosomes or
at the plasma membrane, which leads to the
release of the viral genome into the
cytoplasm [ 66, 67].
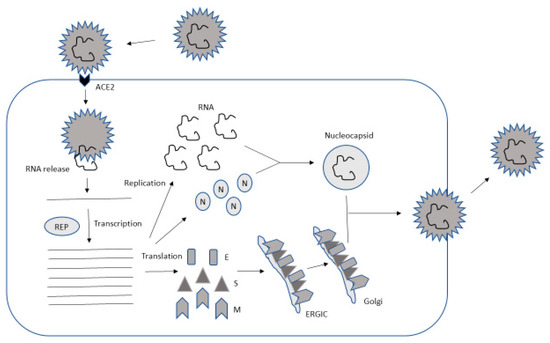
Figure
2. Lifecycle of SARS-CoV-2. The
attachment occurs at ACE2, followed by
release of viral RNA into the cytoplasm.
The replicase (REP) complex is responsible
for RNA replication. RNA and the
translated nucleocapsid (N) protein form
nucleocapsids, while the spike (S),
envelope (E) and membrane (M) proteins go
through the ER–Golgi intermediate
compartment (ERGIC) and Golgi before the
assembly of virus particles takes place on
the plasma membrane from where mature
virions are released.
The translation of the
coronavirus replicase genes from two large
open reading frames (ORFs), rep1a and rep1b,
allows the expression of two polyproteins,
pp1a and pp1ab, utilizing a slippery
sequence (5′-UUUAAAC-3′) and an RNA
pseudoknot [ 68, 69]. The
complicated composition of the pp1a and
pp1ab polyproteins and the assembly of nsPs
are described in detail by Fehr and Perlman
[ 2].
Following the translation and assembly of
the replicase complex, viral RNA synthesis
from both genomic and subgenomic RNAs
occurs. The latter serves as mRNAs for the
structural and accessory genes [ 2]. In the
5′ untranslated region, there are cis-acting
sequences such as stem–loop structures,
which are important for viral RNA
replication [ 70, 71, 72, 73]. An
interesting feature of coronavirus
replication relates to the fusing of the
leader and body TRS segments during the
production of subgenomic RNAs. Although
originally thought to occur during
positive-strand synthesis, a recent model
suggests that RNA-dependent RNA polymerase
(RdRp) pauses at any of the body TRS
sequences during the discontinuous extension
of negative-strand RNA, followed by either
continued elongation to the next TRS or
switching to amplification of the leader
sequence at the 5′ end [ 74, 75].
In the context of
assembly, the viral structural proteins S,
E, and M are translated and transported
along secretory pathways into the
endoplasmic reticulum (ER) and the ER–Golgi
intermediate compartment (ERGIC) [ 76, 77]. At the
ERGIC, viral genomes surrounded by N protein
participate in the formation of mature
virions [ 78].
Assembled virions are then transported to
the cell surface in vesicles and released by
exocytosis [ 79]. An
interesting observation relates to
coronaviruses assembled without the S
protein, which leads to the formation of
giant multi-nucleated cells spreading the
virus within an infected organism without
being detected or neutralized by
virus-specific antibodies [ 2]. 3.
Therapeutic and Prophylactic Options
Since the SARS-CoV
outbreak in 2002–2003, numerous approaches
have been used to develop therapeutic agents
and vaccines for coronaviruses ().
The recent COVID-19 pandemic has further
accelerated the efforts to find cure and
protection. Typically, three general methods
are employed for antiviral treatment of
coronaviruses [ 80]: (i)
application of existing broad-spectrum
antiviral drugs; (ii) screening of chemical
libraries containing many existing compounds
or databases; (iii) redevelopment of new
specific drugs based on the genome and
biophysical understanding of individual
coronaviruses. Other options include
monoclonal antibodies and viral receptor
blockers [ 81].
Products developed as Chinese traditional
medicines have also been suggested [ 80]. In
line with gene therapy applications, the
design of various gene silencing approaches
has been initiated based on RNA interference
(RNAi) [ 82]. In the
context of vaccine development, a wide
variety of immunization strategies have been
initiated [ 83]. 3.1.
Therapeutic Agents
Among therapeutics, the
use of protease inhibitors lopinavir and
ritonavir as a combination therapy with
other antiretroviral agents for the
treatment of HIV-1 infections has provided
durable virologic suppression and improved
immunological outcomes [ 84].
Lopinavir/ritonavir combination treatment
was evaluated in marmosets with a
MERS-like disease [ 85]. In
comparison to mycophenolate mofetil (MFF),
the treatment showed improved clinical,
radiological and pathological outcomes and
lower mean viral loads in necropsied lung
and extrapulmonary tissues, whereas
MFF-treated animals developed severe or
fatal disease. Based on these results, a
study protocol for a clinical trial for
hospitalized adult MERS patients was
prepared [ 86]. The
goal is to investigate
laboratory-confirmed MERS patients in a
recursive, two-stage, multi-center,
placebo-controlled, double-blind
randomized controlled trial. Related to
the recent COVID-19 pandemic, a clinical
trial on 99 patients receiving lopinavir
and ritonavir were compared to 100
patients subjected to standard care.
However, no difference in clinical
improvement, mortality or detectable viral
RNA was obtained [ 87]. In a
modified intention-to-treat analysis, the
median time to clinical improvement was
one day shorter for lopinavir/ritonavir
treatment and although gastrointestinal
adverse events were more common, serious
adverse events were less frequent.
Overall, the study indicated that the
treatment offered no benefit compared to
standard care. Another approach comprises
the application of nucleoside analogues as
antivirals [ 88]. For
instance, several classes of nucleoside
analogues were verified against SARS-CoV
in Vero cells [ 89]. In
the study, the D-isomer of thymine
analogue exhibited strong anti-SARS-CoV
activity and did not show any toxicity at
the highest tested dosage of 100 µM.
Although the L-cytosine analogue showed
good activity, it exhibited strong
toxicity to cells. Likewise, the
3′-azido-2′, 3′-unsaturated thymine
analogue provided strong anti-SARS-CoV
activity, but also significant toxicity.
Additionally, dioxalane-thymine showed
moderate antiviral activity without any
significant cytotoxicity. Although
C-nucleoside,4-amino-7-(β-L-ribofuranosylpyrrolo
[3,2-D] pyrimidine hydrochloride inhibited
SARS-CoV replication, it was cytotoxic.
Overall, despite several classes of
nucleoside analogues against SARS-CoV
exhibiting moderate antiviral activity in
vitro, no clear structure–activity
relationship could be established. In
another study, a series of doubly flexible
nucleoside analogues were designed based
on the acrylic sugar scaffold of acyclovir
[ 90]. One
compound displayed selective antiviral
activity against HCoV-NL63 and MERS-CoV.
In contrast, no activity was detected
against SARS-CoV. In a study on FIP, the
nucleoside analogue GS-441524, a precursor
to the pharmacologically active nucleoside
triphosphate molecule, and acting as an
alternative substrate and RNA chain
terminator of viral RdRp was non-toxic and
effectively inhibited FIPV replication in
feline CRFK cells [ 91].
Moreover, all ten cats treated with
GS-441524 showed rapid reversal of disease
symptoms in two weeks. In another
approach, the guanosine analogue ribavirin
was administered together with
interferon-α2a in adult patients with
laboratory-confirmed MERS-CoV infection
and pneumonia needing ventilation support
[ 92].
The study demonstrated that patients
treated with ribavirin and interferon-α2a
showed a significantly improved survival
rate at 14 days but not at 28 days of
treatment compared to the control group of
patients. The adenosine nucleoside
analogue remdesivir has proven efficient
for inhibition of RNA viruses such as
filoviruses, pneumoviruses and
paramyxoviruses by targeting RdRp [ 93].
Moreover, the antiviral activity of
remdesivir has also been demonstrated for
human endemic and zoonotic coronaviruses
such as HCoV-OC43 and HCoV-229E [ 94]. In
another study, it was shown that
remdesivir potently inhibited RdRp from
MERS-CoV [ 95]. Very
recently, it was demonstrated that
remdesivir effectively inhibited
SARS-CoV-2 in Vero cells and in human
Huh-7 liver cancer cells known to be
susceptible to SARS-CoV-2 [ 96]. The
first case of compassionate treatment of
COVID-19 with intravenous administration
of remdesivir took place in the US,
leading to improvement in the patient’s
condition with a decline in viral load [ 97].
Neuraminidase inhibitors such as
zanamivir, laninamivir, oseltamivir and
peramivir have demonstrated potency
against most influenza strains [ 98]. In
this context, the neuraminidase inhibitors
oseltamivir (Tamiflu) and zanamivir
(Relenza) were tested for inhibition of
cytopathic effects of SARS-CoV in a
cytopathic endpoint assay in Vero cells [ 99].
However, neither neuraminidase inhibitor
showed any effect on SARS-CoV. Arbidol, a
broad-spectrum antiviral agent against
several DNA and RNA viruses, has been
approved in Russia and China for
prevention and treatment of influenza [ 100].
Antiviral effect against Zika virus
(ZIKV), West Nile virus (WNV) and
tick-borne encephalitis virus (TBEV) has
been demonstrated in Vero cells [ 100].
Furthermore, it was shown that arbidol can
inhibit six different isolates of ZIKV and
can protect against the cytopathic effects
of ZIKV [ 101].
Related to SARS-CoV, patients with
laboratory-confirmed COVID-19 were treated
with oral arbidol in combination with
lopinavir/ritonavir or lopinavir/ritonavir
monotherapy [ 102].
After seven days, SARS-CoV could not be
detected in nasopharyngeal specimens in 12
out of 16 patients subjected to the
combination therapy compared to 6 out of
17 for the monotherapy group. The numbers
at day 14 were 15 out of 16 for
combination therapy and 9 out of 17 for
monotherapy at day 14. Furthermore, CT
scans revealed improvement for 11 out of
16 patients receiving the combination
therapy, and 5 out of 17 receiving the
monotherapy.
Table 2. Therapeutic
interventions against coronaviruses.
A hot topic today has
been the potential therapeutic effect of
the widely used antimalarial and
auto-immune disease drug chloroquine on
coronaviruses [ 96].
Chloroquine blocks virus infection and
interferes with the glycosylation of
cellular receptors of SARS-CoV [ 110]. The
application of chloroquine for COVID-19
patients has been justified by its common
use by travelers in malaria-endemic
geographic regions for decades and
continuous use by locals [ 111].
However, as indications of severe side
effects of chloroquine use have been
reported, hydroxychloroquine, possessing a
similar antiviral potency to chloroquine
but a safer clinical profile, should be
considered [ 112]. In
a limited clinical trial, 20 French
COVID-19 patients were treated with
hydroxychloroquine, resulting in a
significant reduction in viral load in
comparison to control patients [ 103].
Addition of azithromycin further
significantly improved the efficiency of
virus elimination. Despite the small
sample size, the study indicated that
hydroxychloroquine treatment was
associated with a significant viral load
reduction and disappearance of COVID-19.
However, the trial design was poor and the
results unreliable, as six patients
dropped out and the assessment of efficacy
was based on viral load, which was not a
clinical endpoint. Additional studies are
therefore needed in a larger number of
patients to thoroughly validate the safety
and efficacy of hydroxychloroquine.
Chinese traditional
medicine has also tried to provide support
in the fight against coronaviruses. In
this context, case studies of treatment
with the Shuanghuanglian oral liquid (SHL)
of a family suffering from COVID-19 has
been reported [ 104].
Case 1, a 51-year-old female with high
fever diagnosed with COVID-19 was confined
into an isolation ward five days later,
where she started to take oral SHL twice a
day. The following day, SHL administration
was increased to three doses a day without
taking any other drugs. The fever
decreased in two days, and she gradually
recovered. Case 2, the 27-year-old
daughter of Case 1 presented with a high
fever, vomiting and diarrhea. Two days
after she was confined in an isolation
ward, she started to take SHL three times
a day. A couple of days later, a decrease
in body temperature and recovered appetite
were noted. Case 3, the husband of Case 1,
had mild diarrhea, vomiting and fever and
was diagnosed with COVID-19. He started to
take SHL three times a day, as well as
moxifloxacin and arbidol, which resulted
in all symptoms with exception of light
nausea disappearing within four days. In
another study, four patients with mild or
severe 2019-nCoV pneumonia were treated
with lopinavir/ritonavir, arbidol and
traditional Chinese medicine in the form
of Shufeng Jiedu capsules [ 105].
Three patients showed significant
improvement related to the pneumonia
symptoms and tested 2019-nCoV negative,
and one patient with severe pneumonia also
showed improvement. Another potential
approach for COVID-19 treatment relates to
AHCC, an α-glucan-based standardized
mushroom extract from Lenintula edodes,
which has demonstrated immunostimulation
in humans infected by WNV, influenza
virus, avian influenza virus, hepatitis C
virus, papilloma virus, herpes virus,
hepatitis B virus and HIV [ 113].
Although the potency of AHCC has not yet
been verified for SARS-CoV-2, it might be
an attractive alternative approach to
explore. Likewise, lianhuaqingwen capsules
have proven to be efficient for the
prevention and treatment of respiratory
infections caused by influenza A [ 114] but
have not yet been validated for
SARS-CoV-2.
In light of therapeutic
strategies to rapidly target SARS-CoV-2,
one approach aims at blocking virus entry
using a soluble version of ACE2 fused to
an immunoglobulin Fc domain [ 81]. This
approach will elicit broad-ranging
neutralizing antibodies and stimulate the
immune system. Recombinant protein
expression can be utilized for the rapid
production of a drug for compassionate use
while formal clinical trials are
undertaken, and vaccines developed.
Another target related to ACE2 comprises
the transmembrane protease serine 2
(TMPRSS2) involved in S protein priming,
for which TMPRSS2 inhibitors might be
approved for clinical use to block
coronavirus entry [ 106]. It
was also demonstrated that sera from
convalescent SARS patients are capable of
cross-neutralizing SARS-CoV-2 S-driven
entry [ 106]. In
the context of ACE2, although the binding
affinity of SARS-CoV and SARS-CoV-2 is
similar, the presence of the furin
cleavage site uniquely in the SARS-CoV-2 S
protein sets it apart for the design of
specific inhibitors [ 57].
Moreover, the finding that SARS-CoV S
murine polyclonal antibodies can potently
prevent SARS-CoV-2 S-mediated cell entry
indicated that cross-neutralizing
antibodies against conserved S epitopes
might be a feasible approach for therapy.
In the context of therapeutic monoclonal
antibodies for SARS-CoV-2, the first
SARS-CoV-specific human monoclonal
antibody (mAb) CR3022 showed potent
binding to the SARS-CoV-2 receptor-binding
domain [ 107].
However, the CR3022 epitope does not
overlap with the ACE2 binding site, which
indicates that CR3022 could have the
potential as a therapeutic as such or in
combination with other neutralizing
antibodies. Interestingly, the potent
SARS-CoV-specific m396 and CR3014
neutralizing antibodies, which target the
ACE2 binding site, failed to bind the
SARS-CoV-2 S protein, suggesting
differences in the receptor-binding
domains of SARS-CoV and SARS-CoV-2. It is
therefore necessary to develop mAbs with
specific binding affinity to the
SARS-CoV-2 receptor-binding domain.
Another tentative approach comprises the
evaluation of existing angiotensin
receptor 1 (ATR1) blockers such as
losartan, commonly used for the treatment
of hypertension [ 108], for
a reduction in aggressiveness and
mortality from SARS-CoV-2 infections [ 115]. The
approach is based on the finding that ACE2
most likely represents the binding site
for both SARS-CoV and SARS-CoV-2, thereby
providing a sensible target for
therapeutic interventions for coronavirus
infections. Targeting ATR1 might also
address the problem with new emerging
coronavirus mutations. Very recently, the
first report on the human 47D11 mAb
targeting a conserved epitope in the spike
receptor-binding domain was published [ 109]. It
cross-neutralizes SARS-CoV and SARS-CoV-2
independently from receptor-binding
inhibition and will be useful for the
development of antigen detection tests and
serological assays. Moreover, it adds to
the potential to prevent and treat
COVID-19 and possible future emerging
coronaviruses. 3.2.
Gene Silencing
Gene silencing based on
RNA interference has proven to be an
important tool in basic research but also
for therapeutic applications [ 116, 117]. The
mechanism of RNAi relates to 19–23 base
pair double-stranded RNAs (dsRNAs)
mediating degradation of target RNA in a
sequence-specific manner [ 118].
Coronaviruses have been subjected to
several gene silencing studies (). For instance, short interfering
RNAs (siRNAs) have been demonstrated to
efficiently inhibit SARS-CoV replication
in Vero E6 cells [ 119].
Moreover, 48 siRNA sequences were designed
throughout the SARS-CoV genome, targeting
several key proteins [ 120].
Chemically synthesized siRNAs were
transfected into fetal rhesus kidney FRhK4
cells before or after SARS-CoV infection
and the inhibitory effects were verified
by the decrease in intracellular viral
genome copy number and viral titers. Four
siRNAs demonstrated potent inhibition of
SARS-CoV infection and replication.
Prophylactic effects with up to 90%
inhibition lasted at least for 72 h.
Combination of siRNA duplexes from
different regions of the viral genome
provided up to 80% inhibition.
Furthermore, siRNA duplexes have been
shown to significantly suppress SARS-like
symptoms in vivo in rhesus macaques [ 121]. In
another approach, expression of U6
promotor-driven siRNA homologous to ACE2
mRNA silenced ACE2 expression in Vero
cells [ 122]. It
was further demonstrated that SARS-CoV
infection was reduced in ACE2-silenced
cell lines, providing an attractive
approach for siRNA-based prophylactic or
therapeutic strategies. Moreover, siRNA
duplexes were applied to knock down
expression of the actin-binding protein
ezrin, which interacts with the SARS-CoV
spike protein during the entry stage of
infection [ 123].
Table 3. Gene silencing against
coronaviruses.
In the context of
MERS-CoV the ORF1ab region encoding the
replicase polyproteins plays a vital role
in viral infection and therefore
represents a suitable target for disease
control. Four miRNA and five siRNA
molecules were rationally designed by
computational methods for silencing of
nine different MERS-CoV strains for
exploration of the treatment of MERS-CoV
at the genomic level [ 124]. In
attempts to optimize delivery of MERS-CoV
siRNAs, transfection, electroporation and
viral gene transfers have been applied.
Recently, advanced nanotechnology based on
lipids, polymers and inorganic compounds
have been formulated [ 128].
Gene silencing of FIPV by siRNAs has been
hampered by mutations creating resistant
viruses. To address the problem,
combination therapy with three siRNAs
prevented viral escape over the course of
five passages [ 125].
Moreover, Dicer-substrate siRNAs provided
equivalent or better potency than
canonical siRNAs for FIPV. In the context
of porcine deltacoronavirus (PDCoV), two
short hairpin RNA (shRNA)-expressing
plasmids targeting the M (pGenesil-M) and
N (pGenesil-N) genes of PDCoV were
evaluated in swine testicular (ST) cells [ 126].
Challenges with the PDCoV HB-BD strain
provided highly specific and efficient
protection of ST cells. Treatment with
pGenesil-M and pGenesil-N resulted in a
13.2- and 32.4-fold titer reduction,
respectively, and a 45.8% and 56.1%
decrease in viral RNA, respectively. In
another study, shRNAs targeting the M gene
of porcine epidemic diarrhea virus (PEDV)
and swine acute diarrhea syndrome
coronavirus (SADS-CoV) and the N gene of
PDCoV inhibited expression of each viral
RNA over 98% [ 127].
Moreover, shRNAs significantly restricted
the expression of M and N proteins and
impaired PEDV, SADS-CoV and PDCoV
replication. 3.3.
Vaccine Development
The classic approach for
vaccine development against viral diseases
has involved immunization with live
attenuated or inactivated viruses [ 129]. The
availability of genetic engineering and
efficient recombinant protein production
technologies has shifted applications to
the utilization of recombinantly expressed
antigens and immunogens for immunization [ 130]. In
the context of coronaviruses, vaccine
development started seriously after the
SARS and MERS outbreaks, providing
alternative approaches of applying subunit
vaccines, whole inactivated virus,
vectored, and live attenuated virus
vaccines [ 83] (). Efforts to tackle other
coronaviruses have also been explored. For
instance, a modified-live vaccine against
BCoV was developed by progressive
attenuation of the respiratory BCoV strain
438/06-TN [ 131]. The
vaccine was proven safe and intramuscular
injection in calves elicited high antibody
titers against BCoV 30 days
post-vaccination. The intranasal drug
Bovilis ® has been approved
against enteric disease caused by BCoV in
young calves [ 132]. The
lack of relevant vaccines for equine
coronavirus (ECoV) catalyzed the study on
antibody responses to ECoV in horses after
vaccination with the BCoV vaccine [ 133].
Antibody titers against ECoV increased in
all six vaccinated horses at 14 days
post-inoculation, although the titers were
lower against ECoV than BCoV, and it
remained unclear whether the elicited
antibodies provided protection against
ECoV.
In the context of
vaccine development, different
computational and informatics tools play
an essential role. For instance, the
immune epitope database (IEDB) has been
used to predict suitable MERS-CoV epitope
vaccines against the most known world
population alleles based on the S and E
proteins [ 134]. The
study showed that highly conserved
sequences in the S and E proteins might be
considered immunogenically protective and
can elicit both neutralizing antibodies
and T cell responses when reacting with B
cells, T helper cell lymphocytes (HTLs)
and cytotoxic T lymphocytes (CTLs). In
another approach, the SARS-CoV-2 S protein
was characterized to obtain immunogenic
epitopes for vaccine development [ 135].
Thirteen major histocompatibility complex
(MHC)-I and three MHC-II epitopes with
antigenic properties were identified. The
epitopes were linked by specific linkers
and docked to toll-like receptor-5 (TLR5),
and immunoinformatics analysis was
utilized for fast immunogenic profiling to
accelerate vaccine development. In another
immunoinformatics and computational
approach, conserved B and T cell epitopes
for the MERS-CoV S protein were identified
[ 136]. The
antigenicity of the epitopes and
interactions with the human leukocyte
antigen (HLA) B7 allele were estimated.
The highest antigenicity score was
obtained for the immensely immunogenic B
cell epitope QLQMGFGITVQYGT. The T cell
epitope peptides YKLQPLTFL (MHC-I) and
YCILEPRSG (MHC-II) were also highly
antigenic. These identified putative
antigenic epitopes may prove effective for
the development of novel vaccines. Using
in silico approaches, two multi-epitope
vaccines against MERS-CoV were designed by
screening CTL and HTL epitopes from 13
different MERS-CoV proteins [ 137].
Both multi-epitope vaccines also carried
potential B cell linear epitope regions, B
cell discontinuous epitopes and
interferon-γ-inducing epitopes. Moreover,
human β-defensin-2 and β-defensin-3 were
used as adjuvants for enhanced immune
responses. The most potent CTL and HTL
epitopes and adjuvants were linked by
short peptide molecular linkers. Tertiary
models for both multi-epitope vaccines
were verified for their molecular
interaction with TLR3 and cDNAs were
generated for in silico analysis of
expression in human cell lines before
being tested in vivo as potential vaccine
candidates. Computational approaches
and immunoinformatics have provided strong
support for vaccine development as
described above. Moreover,
structure–function studies have also
contributed to the field. In this context,
in comparison to antibodies targeting the
receptor-binding domain on the MERS-CoV S
protein, less attention has been paid to
antibodies targeting non-receptor-binding
domain epitopes such as the neutralizing
antibody G2, which targets the MERS-CoV
N-terminal domain of S1 [ 138].
Structural and functional characterization
of G2 alone or complexed with the MERS-CoV
N-terminal of S1 demonstrated that G2
strongly disrupts the attachment of
MERS-CoV S to the DDP4 receptor and could
play an important role as a target for
immunotherapy and vaccine development. In
another approach, cryo-electron microscopy
(cryo-EM) was applied for the human
HCoV-NL63, providing a 3.4 Å resolution of
the spike glycoprotein trimer essential
for viral entry into host cells and
representing the main target for
neutralizing antibodies [ 139]. The
structure revealed important components of
the fusion process including the
triggering loop and the C-terminal domains
involved in the anchoring of the trimer to
the viral membrane. The study also
revealed that HCoV-NL63 use molecular
trickery based on epitope masking with
glycans and activating conformational
changes in attempts to evade recognition
by the host immune system. In another
study, the SARS-CoV-2 S protein was
subjected to cryo-EM structure
determination of the ectodomain trimer for
the identification of potential targets
for vaccines and viral entry inhibition as
the furin cleavage site between the S1 and
S1 subunits is unique for SARS-CoV-2 and
not present in SARS-CoV or other related
coronaviruses [ 57]. In a
study on SARS-CoV, screening of
experimentally determined B and T
cell-derived epitopes from the S and N
proteins, which maps identically to
SARS-CoV-2, showed no mutations among the
120 available SARS-CoV-2 sequences [ 140].
Immune targeting of these epitopes may
therefore potentially offer protection
against SARS-CoV-2. Related to the T cell
epitopes, a population coverage analysis
of associated MHC alleles was conducted,
allowing the identification of a set of
epitopes, which might provide a broad
global coverage.
Table 4. Vaccine approaches
against voronaviruses.
Nucleic acid-based
vaccines have become attractive
alternatives to vaccines based on live
attenuated or inactivated viruses. In this
context, four plasmid DNA-based vaccine
constructs were intradermally administered
into C57BL/6 mice [ 141]. The
pLL70 vector contained the SARS-CPoV S
gene and the pcDNA-SS vector contained the
codon-optimized SARS-CoV S gene fused with
the leader sequence from the human CD5
gene. The pcDNA-St vector carried the
N-portion of the codon-optimized S gene
with the CD5 leader sequence and the
pcDNA-St-VP22C contained the N-portion of
the codon-optimized S with the CD5 leader
sequence fused to the C-terminal of the
bovine herpesvirus-1 (BHV-1) VP22 protein,
known to facilitate and enhance protein
delivery [ 156].
Immunization studies revealed that
pcDNA-SS and pcDNA-St-VP22C elicited
superior cellular and humoral immune
responses in mice and therefore represent
the most immunogenic SARS vaccine
candidates. Moreover, the DNA vaccine
approach was verified targeting the
SARS-CoV S protein using an improved
plasmid DNA vector containing donor and
acceptor splice sites and heterologous
viral RNA export elements such as the
constitutive transport element (CTE) and
the post-transcriptional regulatory
element (PRE) from Mason-Pfizer monkey
virus and Woodchuck hepatitis virus,
respectively [ 142].
These vector modifications significantly
improved the immunogenicity and
immunization of mice with 2 µg of naked
DNA induced neutralizing anti-S antibodies
and provided protection against challenges
with SARS-CoV.
In another plasmid
DNA-based study, immune responses to a
naked plasmid vector and
plasmid/polyethylenimine nanoparticles
expressing the SARS-CoV S protein were
investigated in BALB/c mice after
intranasal administration [ 143].
Immunization with nanoparticles elicited
significantly higher S-specific IgG1
antibodies in sera and mucosal secretory
IgA antibodies in lung wash than in mice
receiving naked plasmid DNA. In another
study, DNA plasmid-based prime
immunization was combined with a boost
vaccination with a protein [ 144].
Turkeys were immunized with one or two
doses of 750 μg of a DNA plasmid carrying
a turkey coronavirus (TCoV) S protein
fragment containing neutralizing epitopes
(4F/4R) followed by a boost with 200 μg of
4F/4R fragment. Animals were challenged
with infectious TCoV and clinical signs
were monitored by an immunofluorescence
antibody assay. Immunized turkeys showed
less clinical signs and a lower viral load
compared to control animals. The
vaccination also induced humoral immune
responses and provided partial protection
against challenges with TCoV.
Cellular and viral
vector-based expression systems play an
important role in vaccine development. The
large size of the CoV genome has
complicated the construction of infectious
clones for utilization in studies on basic
viral processes and development of
genetically defined vaccines [ 156]. In
this context, bacterial artificial
chromosomes (BACs) can provide a robust
system for expression of viral RNA in the
nucleus under the control of a
cytomegalovirus (CMV) promoter followed by
RNA amplification by the viral replicase
in the cytoplasm [ 157]. In
a prokaryotic approach, a eukaryotic
recombinant plasmid expressing the SAD
epitope (A and D antigenic sites of the S
protein) of TGEV was transformed into Lactobacillus
acidophilus originating from
swine [ 145].
Oral administration of L. acidophilus
in BALB/c mice induced significantly
higher levels of S IgA antibodies compared
to a commercial inactivated TGEV vaccine.
The levels of TGEV-specific IgGs were
similar but higher levels of interferon-γ
were induced by the L. acidophilus
vaccine. Overall, the oral TGEV L. acidophilus
vaccine induced high levels of both
mucosal and humoral immune responses. In
another approach, a stable CHO cell line
was engineered to express a subunit
recombinant protein vaccine of residues
377-588 of the receptor-binding domain of
the MERS-CoV S protein [ 146]. The
vaccine has been demonstrated to elicit
significant neutralizing antibody
responses and can provide protection
against MERS-CoV challenges in vaccinated
animals. For stable expression, the IL-2
signal peptide was introduced in front of
the S protein domain fused to the human
IgG Fc fragment and transfected into an
adherent dihydrofolate reductase-deficient
CHO cell line. The adCHO-expressed fusion
protein was secreted and showed
functionality and binding specificity, and
a suspension CHO cell line has been
developed. Engineered transgenic mice with
the DPP4 receptor susceptible to MERS-CoV
were immunized with the S377-588-Fc
subunit vaccine and adjuvant, which
resulted in production of neutralizing
antibodies against MERS-CoV and survival
for at least 21 days after challenges with
live MERS-CoV.
An interesting approach
has been to utilize plant expression
systems for coronavirus vaccine
development. For instance, the N-terminal
part (amino acids 1-658) of the SARS-CoV S
protein was optimized for codon usage in
plants and expressed as a fusion protein
with the green fluorescent protein (GFP)
in tobacco leaves [ 147]. It
was demonstrated that the S1-GFP fusion
protein was expressed in the cytoplasm.
Stable expression from the cauliflower
mosaic virus 35S promoter resulted in a
high level of expression of the fusion
protein in tobacco and lettuce leaves.
Moreover, S1 production was also achieved
in chloroplast-transformed plants,
suggesting the potential for developing
safe oral plant-derived subunit vaccines
against SARS-CoV. In another study, an
N-terminal fragment of the SARS-CoV S
protein was expressed at high levels in
tomato and low-nicotine tobacco plants [ 148]. The
plant-derived antigen elicited systemic
and mucosal immune responses in mice.
Significantly increased levels of
SARS-CoV-specific IgA antibodies were
detected in mice after oral administration
of tomato fruits expressing the S1
protein. Moreover, SARS-CoV-specific IgG
antibodies were detected in the serum of
mice primed with tobacco-derived S1
protein. Enhanced expression of the
SARS-CoV N protein was achieved in the
tobacco plant Nicotiana
benthamiana by including the
post-transcriptional gene silencing
suppressor p19 protein from tomato bushy
stunt virus [ 149].
Intraperitoneal administration of plant
extract in BALB/c mice elicited N
protein-specific IgG antibodies and,
overall, it could be concluded that
plant-based expression of the SARS-CoV N
protein can induce strong humoral and
cellular immune responses in mice.
Finally, recent progress in plant-based
expression systems has allowed the
production of numerous antigens and
monoclonal antibodies in plants [ 158],
which has now also accelerated the
application of plant-based expression
systems for rapid vaccine development
against SARS-CoV-2.
Viral vectors have been
frequently used as delivery vehicles for
immunization against infectious agents
such as pathogenic viruses [ 159]. In
many cases, strong humoral and cellular
immune responses have been observed as
well as protection against challenges with
lethal doses of pathogenic viruses. In the
context of coronaviruses, a recombinant
adenovirus vector expressing the
heat-labile enterotoxin B (LTB) and the
core neutralizing epitope (COE) of PEDV
was administered intramuscularly or orally
into BALB/c mice and piglets [ 150].
Three vaccinations at two-week intervals
generated robust humoral and cellular
immune responses. Cell-mediated immune
responses were seen in mice and
neutralizing antibodies inhibited both the
vaccine strain and emerging PEDV isolates.
Strong immune responses were observed in
piglets, but further studies are required
to verify the protection against
challenges with highly virulent PEDV
strains. In another study, the modified
vaccinia virus Ankara (MVA) strain was
utilized for the expression of recombinant
MERS-CoV S protein [ 151].
Immunization of dromedary camels elicited
mucosal immunity. Immunized camels showed
significantly reduced excretion of
infectious virus and viral RNA transcripts
and protection against MERS-CoV correlated
with the presence of neutralizing
antibodies in the serum. In another
application of MVA, the MERS-CoV N was
used for induction of cellular immune
responses [ 152].
Identification of MHC-I- and
MHC-II-restricted T cell responses was
carried out on overlapping peptides
spanning the whole MERS-CoV N polypeptide
in BALB/c mice immunized with MVA-MERS-CoV
N. An H2-d restricted decamer peptide
epitope showing CD8 + T cell
antigenicity was identified, which will be
further subjected to protection studies in
mouse models for MERS-CoV. In the context
of SARS-CoV, the N and S protein genes
were cloned between the glycoprotein G and
polymerase L genes in a highly attenuated
safe rabies virus (RV) vector [ 153]. A
single injection with the RV-SARS-CoV S
vector induced strong neutralizing
antibody responses in mice, which makes it
a promising candidate for eradication of
SARS-CoV from animal reservoirs.
Finally,
self-replicating RNA viruses have proven
to be efficient as vaccine vectors due to
the highly efficient replication of their
ssRNA genome of positive polarity directly
in the cytoplasm [ 159]. As
vectors have been engineered allowing
immunization of plasmid DNA replicons,
naked RNA replicons or
replication-deficient particles, the
application range is wide. Moreover, due
to self-replication, the amounts required
for obtaining similar immune responses as
seen for conventional DNA plasmids or
synthetic mRNA molecules are 100- to
1000-fold lower [ 160]. In
the context of coronaviruses, Venezuelan
equine encephalitis virus (VEE) replicon
particles were used for the expression of
the Urbani SARS-CoV S or N proteins [ 154]. It
was demonstrated that VEE particles
expressing SARS-CoV S but not N provided
complete short- and long-term protection
against challenges with homologous strains
in both young and senescent mice. To
evaluate challenges of heterologous
strains, a chimeric virus (icGDO3-S)
encoding a synthetic S gene of the most
genetically divergent human GDO3 strain
was engineered. The chimeric virus was
highly resistant to neutralization with
antisera directed against the Urbani
strain. However, immunization with VEE
particles expressing SARS-CoV S provided
complete short-term protection against
challenges with icGDO3-S in young mice,
but not in senescent mice. The failure to
protect senescent mice was addressed in
another study, where mice were vaccinated
with VEE particles expressing SARS-CoV S
antigen packaged with either attenuated
(3014) or wild-type (3000) VEE
glycoproteins [ 155]. The
results revealed that aged animals
immunized with VEE (3000)-based vaccine
were protected against SARS-CoV, while
mice immunized with VEE (3014)-based
vaccine were not. Furthermore, the
superior protection was also confirmed for
challenges with influenza virus.
Related to the safety of
vaccine development, antibody-dependent
enhancement (ADE) comprises an important
mechanism facilitating viral cell entry,
in which virions are complexed with
antibodies, resulting in enhanced viral
replication [ 161]. In
the context of COVID-19, prior infection
with other coronaviruses, from common cold
to SARS-CoV, may have primed COVID-19
patients, causing severe disease once
infected with SARS-CoV-2. This could
explain the discrepancy between the
severity of disease in COVID-19 patients
in the Hubei province in China compared to
other regions of the world [ 162].
Recently, a novel molecular mechanism for
ADE was revealed, demonstrating that a
neutralizing antibody binding to the
coronavirus S protein can trigger a
conformational change of the S protein
mediating viral entry via IgG Fc cellular
receptors [ 163].
Another issue related to vaccine
development comprises the type II cellular
immunity for lung pathogenicity. In this
context, accumulated evidence suggests
that the lungs are a major site of immune
regulation [ 164].
Therefore, a highly regulated immune
response in the lungs can protect from
pathogen infection. In contrast,
inefficient immune responses can trigger
various pulmonary diseases. Related to
SARS and COVID-19 lung pathology, recovery
requires rigorous innate and acquired
immune responses and epithelial
regeneration [ 165].
However, administration of epithelial
growth factors such as the keratinocyte
growth factor (KGF) might stimulate the
production of ACE2-expressing cells,
increasing the viral load.
An immunotherapeutic
approach of great interest relates to the
use of plasma from convalescent COVID-19
patients. Convalescent plasma has
previously been used successfully as
post-exposure prophylaxis and/or treatment
of SARS and MERS [ 166]. In
the context of COVID-19, in a case study,
a patient with severe COVID-19 was treated
with convalescent plasma from six donors [ 167]. The
anti-SARS-CoV IgM responses from the
convalescent plasma were weak, but high
titers of IgG were obtained. The treatment
allowed the patient to be released from
mechanical ventilation 11 days after
plasma transfusion and then transferred to
a general ward. In another case study,
five critically ill COVID-19 patients with
acute respiratory distress syndrome (ARDS)
were treated with convalescent plasma
obtained from patients who had recovered
from COVID-19 [ 168]. The
patients who were receiving mechanical
ventilation and had been treated with
antiviral agents and methylprednisolone
showed a normalized body temperature
within 3–4 days and their viral load
decreased and became negative within 12
days. Moreover, SARS-CoV-2-specific ELISA
and neutralizing antibody titers
increased, ARDS resolved in four patients
after 12 days and three patients did not
need mechanical ventilation after two
weeks. Three patients were discharged from
the hospital and the two remaining
patients were in stable condition.
Obviously, the limited number of patients
and study design do not permit an
evaluation of the efficacy of the
treatment and further larger clinical
trials are required. 4.
Conclusions and Future Aspects
In summary, there are no
efficient antiviral drugs or vaccines
currently available for COVID-19. However,
due to the extent of the current COVID-19
pandemic, it seems like the whole world has
come together to conquer the outbreak. There
seems to be enormous political and economic
will to support research and development
efforts in an unprecedented way. The
positive outcome is that all avenues are
explored including antiviral drugs in the
form of existing drugs for other viral
diseases, which should be subjected to
thorough well-planned clinical evaluation.
Moreover, new drugs targeting SARS-CoV
receptors in the form of small molecules and
monoclonal antibodies and gene silencing
approaches preventing SARS-CoV replication
are being explored.
As drug development and
particularly antiviral drugs used for other
indications have seen a renaissance of
clinical trials for COVID-19, it is
appropriate to summarize recent clinical
findings. In this context, remdesivir was
applied for compassionate use in 53
hospitalized COVID-19 patients—of which, 22
were in the US, 22 in Europe or Canada and 9
in Japan [ 169].
Thirty-six of the 53 patients (68%) showed
clinical improvement, 25 patients were
discharged and 7 died. Additionally,
COVID-19 patients were subjected to a
clinical trial for remdesivir in China [ 170]. Of
the 237 patients enrolled in the study, 158
received remdesivir and 79 were in the
placebo group. The study indicated that
Remdesivir was not associated with
statistically significant clinical benefits
although numerical reduction in time to
clinical improvement was observed. On 29
April 2020, Gilead, the manufacturer of
remdesivir, put out a press release on
preliminary data on their phase III SIMPLE
trials in COVID-19 patients [ 171]. The
first of the two randomized, open-label,
multicenter SIMPLE trials in 397 patients
demonstrated that the time of clinical
improvement for 50% of patients was 10 days
in the group receiving intravenous
remdesivir for 5 days and 11 days after 10
days of treatment compoared to 15 days for
the control group and more than half of the
patients in both groups were discharged from
the hospital by day 14. The plan is to
extend the study by enrolling an additional
5600 patients and conducting trials at 180
sites in China, France, Germany, Hong Kong,
Italy, Japan, Korea, the Netherlands
Singapore, Spain, Sweden, Switzerland,
Taiwan, the United Kingdom and the United
States. In the second SIMPLE trial, the
safety and efficacy of 5- and 10-day dosing
duration of intravenous remdesivir will be
compared to standard of care treatment of
COVID-19 patients with the first results
expected by the end of May 2020.
Regarding
hydroxychloroquine, initial clinical
evaluation in 20 patients was conducted in
France, as described earlier [ 106].
Although the study indicated some
therapeutic effect of hydroxychloroquine and
at least 80 trials on chloroquine,
hydroxychloroquine, or their combination
with other drugs have been registered
globally, one should be cautious, as many
proposals are based on in vitro studies,
animal models or experiences from other
viral diseases [ 172]. For
instance, a Chinese trial in more than 100
patients demonstrated superiority of
chloroquine phosphate compared to control
treatment in inhibition of pneumonia
exacerbation [ 173].
Moreover, in a placebo-controlled randomized
trial of two different doses of
hydroxychloroquine in 62 patients with
radiological conformation of pneumonia, but
without severe hypoxia, small improvements
in body temperature and cough were
registered only for the higher dose [ 174].
However, the results from the lower dose
were not described, the endpoints specified
in the published protocol differed from
those reported and the trial was terminated
prematurely [ 175].
Supporters of hydroxychloroquine have
referred to the history of wide and safe use
of the drug. However, hydroxychloroquine
will potentially expose patients to serious
cutaneous adverse reactions [ 176],
hepatic failure [ 177] and
ventricular arrhythmias when co-administered
with azithromycin [ 178].
In the context of
lopinavir/ritonavir, a randomized,
controlled, open-label trial conducted on
199 COVID-19 patients showed no difference
related to clinical improvement compared to
the standard-care control group [ 87].
Detectable viral RNA levels and mortality
numbers were similar for the two groups.
Gastrointestinal adverse events were more
common for patients treated with
lopinavir/ritonavir, but serious adverse
events were more frequent in the
standard-care group. The Efficacy of
Lopinavir Plus Ritonavir and Arbidol against
Coronavirus Infection (ELACOI) single-blind
randomized controlled trial in China
included 44 patients with mild or moderate
clinical status taking lopinavir/ritonavir
or arbidol (Umifenovir) [ 179]. The
study showed no differences in the time to
negative pharyngeal SARS-CoV-2 PCR
detection, pyrexia, cough or lung CT
findings between the treatment and control
groups. However, in the lopinavir/ritonavir
group, 38.1% of patients deteriorated to
severe/critical status compared to 12.5% and
14.3% for the arbidol and control groups,
respectively. No adverse events were
registered in the arbidol and control
groups. In contrast, gastrointestinal and
deranged liver adverse events were seen
after lopinavir/ritonavir treatment.
Overall, although drugs
developed for other infectious diseases
might provide alternative treatment
strategies for COVID-19 when evaluated in
well-designed and well-executed clinical
trials, development of prophylactic drugs
and vaccines targeting specific structures
of SARS-CoV-2 seems like the preferred
approach. In the context of vaccine
development, utilization of purified epitope
peptides and antigens and cellular or viral
delivery systems should all be explored in
parallel to achieve maximum success in a
minimum time frame. Not surprisingly, more
than 60 vaccine projects are in progress at
pre-clinical or clinical levels [ 180]. These
initiatives apply all possible means of
delivery including mRNA, plasmid DNA,
non-replicating viral vectors, inactivated
and live attenuated virus and protein
subunits.
It will also be necessary
to look ahead to the possibility of
preventing SARS-CoV-2 outbreaks in the
future and, if not, to be better prepared
for a second or third wave of coronavirus or
any other pandemic. It is important to
understand the origin of SARS-CoV-2 rather
than place blame on bats for the cause of
the pandemic [ 181]. On
several occasions, it has been pointed out
that bats are the only flying mammals. How
wrong is this? In fact, human beings are the
real flying mammals, jetting around the
world at an ever-accelerating pace and
frequency. Only last year, an estimated 4.5
billion passengers took to the skies.
Furthermore, in defense of bats, recent
findings suggest that pangolins are the
prime suspects as the source of SARS-CoV-2
although it has yet to be confirmed [ 182]. In
any case, all trails lead back to humans, as
pangolins are sought after for their meat
and scales. Instead of the blame game, the
focus should now be on accelerated efforts
to develop novel safe and efficacious
prophylactic and therapeutic approaches in
the form of coronavirus drugs and vaccines.
Funding
This research received no
external funding.
Conflicts
of Interest
The author declares no
conflict of interest.
References
- De Groot,
R.J.; Baker, S.C.; Baric, R.; Enjuanes,
L.; Gorbalenya, A.E.; Holmes, K.V.;
Perlman, S.; Poon, L.; Rottier, P.J.;
Talbot, P.J.; et al. Family Coronaviridae.
In Ninth Report
of the International Committee on
Taxonomy of Viruses;
International Committee on Taxonomy of
Viruses, International Union of
Microbiological Societies. Virology
Division; King, A.M., Lefkowitz, E.,
Adams, M.J., Carstens, E.B., Eds.;
Elsevier: Oxford, UK, 2011; pp. 806–828.
ISBN 978-0-12-384684-6. [Google Scholar]
- Fehr,
A.R.; Perlman, S. Coronaviruses: An
Overview of Their Replication and
Pathogenesis. Methods
Mol. Biol. 2015, 1282, 1–23. [Google Scholar]
[PubMed]
- Doyle,
L.P.; Hutchings, L.M. A transmissible
gastroenteritis in pigs. J. Am. Vet. Med.
Assoc. 1946, 108, 257–259.
[Google Scholar]
[PubMed]
- Lin,
C.M.; Saif, L.J.; Marthaler, D.; Wang, Q.
Evolution, antigenicity and pathogenicity
of porcine epidemic diarrhea virus. Virus Res. 2016,
226,
20–39. [Google Scholar]
[CrossRef]
[PubMed]
- Mora-Díaz,
J.C.; Piñeyro, P.E.; Houston, E.;
Zimmerman, J.; Giménez-Lirola, L.G.
Porcine Hemagglutinating Encephalomyelitis
Virus: A Review. Front. Vet. Sci.
2019, 6,
53. [Google Scholar]
[CrossRef]
- Amer,
H.M. Bovine-like coronaviruses in domestic
and wild ruminants. Anim. Health Res.
Rev. 2018, 19, 113–124.
[Google Scholar]
[CrossRef]
- Perlman,
S.; Netland, J. Coronaviruses post-SARS:
Update on replication and pathogenesis. Nat. Rev. Microbiol.
2009, 7,
439–450. [Google Scholar]
[CrossRef]
- Funk,
C.J.; Manzer, R.; Miura, T.A.; Groshong,
S.D.; Ito, Y.; Travanty, E.A.; Leete, J.;
Holmes, K.V.; Mason, R.J. Rat respiratory
coronavirus infection: Replication in
airway and alveolar epithelial cells and
the innate immune response. J. Gen. Virol.
2009, 90,
2956–2964. [Google Scholar]
[CrossRef]
- Cavanagh,
D. Coronavirus avian infectious bronchitis
virus. Vet.
Res. 2007, 38, 281–297.
[Google Scholar]
[CrossRef]
- Wolfe,
L.G.; Griesemer, R.A. Feline infectious
peritonitis. Pathol.
Vet. 1966, 3, 255–270. [Google Scholar]
[CrossRef]
- Jaimes,
J.A.; Whittaker, G.A. Feline coronavirus:
Insights into viral pathogenesis based on
the spike protein structure and function.
Virology
2018, 517,
108–121. [Google Scholar]
[CrossRef]
- Izumi,
T. Sarcoidosis in various organs. Clin. Dermatol.
1986, 4,
150–153. [Google Scholar]
[CrossRef]
- Mihindukulasuriya,
K.A.; Wu, G.; St Leger, J.; Nordhausen,
R.W.; Wang, D. Identification of a novel
coronavirus from a beluga whale by using a
panviral microarray. J. Virol. 2008,
82,
5084–5088. [Google Scholar]
[CrossRef]
[PubMed]
- He, B.;
Zhang, Y.; Xu, L.; Yang, W.; Yang, F.;
Feng, Y.; Xia, L.; Zhou, J.; Zhen, W.;
Feng, Y.; et al. Identification of diverse
alphacoronaviruses and genomic
characterization of a novel severe acute
respiratory syndrome-like coronavirus from
bats in china. J.
Virol. 2014, 88,
7070–7082. [Google Scholar]
[CrossRef]
[PubMed]
- Menachery,
V.D.; Yont, B.L., Jr.; Debbink, K.;
Agnihothram, S.; Gralinski, L.E.; Plante,
J.A.; Graham, R.L.; Scobey, T.; Ge, X.Y.;
Donaldson, E.F.; et al. A SARS-like
cluster of bat coronaviruses shows
potential for human emergence. Nat. Med. 2015,
21,
1508–1513. [Google Scholar]
[CrossRef]
- Wege,
H.; Siddell, S.G.; ter Meulen, V. The
biology and pathogenesis of coronaviruses.
Curr. Top
Microbiol. Immunol. 1982,
99,
165–200. [Google Scholar]
- Lampert,
P.W.; Sims, J.K.; Kniazeff, A.J. Mechanism
of demyelination in JHM virus
encephalomyelitis. Acta Neuropathol.
1973, 24,
76–85. [Google Scholar]
[CrossRef]
- Bradburne,
A.F.; Bynoe, M.L.; Tyrell, D.A.J. Effects
of a “new” human respiratory virus in
volunteers. Br.
Med. J. 1967, 3, 767–769. [Google Scholar]
[CrossRef]
- van der
Hoek, L.; Sure, K.; Ihorst, G.; Stang, A.;
Pyrc, K.; Jebbink, M.F.; Petersen, G.;
Forster, J.; Berkhout, B.; Uberla, K.
Croup is associated with the novel
coronavirus NL63. PLoS Med. 2005,
2, e240.
[Google Scholar]
[CrossRef]
- Hamre,
D.; Procknow, J.J. A new virus isolated
from the human respiratory tract. Proc. Soc. Exp.
Biol. Med. 1966, 121, 190–193.
[Google Scholar]
[CrossRef]
- Anderson,
R.M.; Fraser, C.; Ghani, A.C.; Donelly,
C.A.; Riley, S.; Ferguson, N.M.; Leung,
G.M.; Lam, T.H.; Hedley, A.J.
Epidemiology, transmission dynamics and
control of SARS: The 2002–2003 epidemic. Philos. Trans. R
Soc. London B Biol. Sci. 2004,
359,
1091–1105. [Google Scholar]
[CrossRef]
- Cherry,
J.D. The chronology of the 2002–2003 SARS
mini pandemic. Paediatr.
Resp. Rev. 2004, 5, 262–269. [Google Scholar]
[CrossRef]
[PubMed]
- Aleanizi,
F.S.; Mohmed, N.; Alqahtani, F.Y.; El Hadi
Mohamed, R.A. Outbreak of Middle east
respiratory syndrome coronavirus in Saudi
Arabia: A retrospective study. BMC Infect. Dis.
2017, 17,
23. [Google Scholar]
[CrossRef]
[PubMed]
- Guan,
Y.; Zheng, B.J.; He, Y.Q.; Liu, X.L.;
Zhuang, Z.X.; Cheung, C.L.; Luo, S.W.; Li,
P.H.; Zhang, L.J.; Guan, Y.J.; et al.
Isolation and characterization of viruses
related to the SARS coronavirus from
animals in southern China. Science 2003,
302,
276–278. [Google Scholar]
[CrossRef]
[PubMed]
- Lau,
S.K.; Woo, P.C.; Li, K.S.; Huang, Y.;
Tsoi, H.W.; Wong, B.H.; Wong, S.S.; Leung,
S.Y.; Chan, K.H.; Yuen, K.Y. Severe acute
respiratory syndrome coronavirus-like
virus in Chinese horseshoe bats. Proc. Natl. Acad.
Sci. USA 2005, 102,
14040–14045. [Google Scholar]
[CrossRef]
- Li, W.;
Shi, Z.; Yu, M.; Ren, W.; Smith, C.;
Epstein, J.H.; Wang, H.; Crameri, G.; Hu,
Z.; Zhang, H.; et al. Bats are natural
reservoirs of SARS-like coronaviruses. Science 2005,
310,
676–679. [Google Scholar]
[CrossRef]
- Ge,
X.Y.; Li, J.L.; Yang, X.L.; Chmura, A.A.;
Zhu, G.; Epstein, J.H.; Mazet, J.K.; Hu,
B.; Zhang, W.; Peng, C.; et al. Isolation
and characterization of a bat SARS-like
coronavirus that uses the ACE2 receptor. Nature 2013,
503,
535–538. [Google Scholar]
[CrossRef]
- Peiris,
J.S.; Yuen, K.Y.; Osterhaus, A.D.; Stohr,
K. The severe acute respiratory syndrome.
N. Engl. J. Med.
2003, 349,
2431–2441. [Google Scholar]
[CrossRef]
- Zaki,
A.M.; van Boheemen, S.; Bestebroer, T.M.;
Osterhaus, A.D.; Fouchier, R.A. Isolation
of a novel coronavirus from a man with
pneumonia in Saudi Arabia. N. Engl. J. Med.
2012, 367,
1814–1820. [Google Scholar]
[CrossRef]
- van
Boheemen, S.; de Graaf, M.; Lauber, C.;
Bestebroer, T.M.; Raj, V.S.; Zaki, A.M.;
Osterhaus, A.D.; Haagmans, B.L.;
Gorbalenya, A.E.; Snijder, E.J.; et al.
Genomic characterization of a newly
discovered coronavirus associated with
acute respiratory distress syndrome in
humans. mBio
2012, 3,
e00473-12. [Google Scholar]
[CrossRef]
- Chibo,
D.; Birch, C. Analysis of human
coronavirus 229E spike and nucleoprotein
genes demonstrates genetic drift between
chronologically distinct strains. J. Gen. Virol.
2006, 87,
1203–1208. [Google Scholar]
[CrossRef]
- Vijgen,
L.; Keyaerts, E.; Lemey, P.; Moes, E.; Li,
S.; Vandamme, A.M.; Van Ranst, M.
Circulation of genetically distinct
contemporary human coronavirus OC43
strains. Virology
2005, 337,
85–92. [Google Scholar]
[CrossRef]
[PubMed]
- Meyer,
B.; Muller, M.A.; Corman, V.M.; Reusken,
C.B.; Ritz, D.; Godeke, G.J.; Lattwein,
E.; Kallies, S.; Siemens, A.; van Beek,
J.; et al. Antibodies against MERS
coronavirus in dromedary camels, United
Arab Emirates, 2003 and 2013. Emerg. Infect. Dis.
2014, 20,
552–559. [Google Scholar]
[CrossRef]
[PubMed]
- Eckerle,
I.; Corman, V.M.; Muller, M.A.; Lenk, M.;
Ulrich, R.G.; Drosten, C. Replicative
Capacity of MERS Coronavirus in Livestock
Cell Lines. Emerg.
Infect. Dis. 2014, 20, 276–279.
[Google Scholar]
[CrossRef]
[PubMed]
- Azhar,
E.I.; El-Kafrawy, S.A.; Farraj, S.A.;
Hassan, A.M.; Al-Saeed, M.S.; Hashem,
A.M.; Madani, T.A. Evidence for
Camel-to-Human Transmission of MERS
Coronavirus. N.
Engl. J. Med. 2014, 370,
2499–2505. [Google Scholar]
[CrossRef]
- Raj,
V.S.; Mou, H.; Smits, S.L.; Dekkers, D.H.;
Muller, M.A.; Dijkman, R.; Muth, D.;
Demmers, J.A.; Zaki, A.; Fouchier, R.A.;
et al. Dipeptidyl peptidase 4 is a
functional receptor for the emerging human
coronavirus-EMC. Nature 2013,
495,
251–254. [Google Scholar]
[CrossRef]
- Zhao,
J.; Li, K.; Wohlford-Lenane, C.;
Agnihothram, S.S.; Fett, C.; Zhao, J.;
Gale, M.J., Jr.; Baric, R.S.; Enjuanes,
L.; Gallagher, T.; et al. Rapid generation
of a mouse model for Middle East
respiratory syndrome. Proc. Natl. Acad.
Sci. USA 2014, 111,
4970–4975. [Google Scholar]
[CrossRef]
- Kaplan,
E.H. Containing 2019-nCoV (Wuhan)
coronavirus. Health
Care Manag. Sci. 2020. [Google Scholar]
[CrossRef]
- Yang,
Y.; Peng, F.; Wang, R.; Guan, K.; Jiang,
T.; Xu, G.; Sun, J.; Chang, C. The deadly
coronavirus; The 2003 SARS epidemic and
the 2020 novel coronavirus epidemic in
China. J.
Autoimmun. 2020. [Google Scholar]
[CrossRef]
- Global
Health Policy. Available online: https://www.google.com/covid19-map
(accessed on 28 April 2020).
- Han,
Q.; Lin, Q.; Jin, S.; You, L. Coronavirus
2019-nCoV: A brief perspective from the
front line. J.
Infect. 2020. [Google Scholar]
[CrossRef]
- Kahn,
J.; McIntosh, K. History and recent
advances in coronavirus discovery. Ped. Infect. Dis. J.
2005, 24,
s223–s227. [Google Scholar]
[CrossRef]
- Zhao,
L.; Jha, B.K.; Wu, A.; Elliott, R.;
Ziebuhr, J.; Gorbalenya, A.E.; Silverman,
R.H.; Weiss, S.R. Antagonism of the
interferon-induced OAS-RNase L pathway by
murine coronavirus ns2 protein is required
for virus replication and liver pathology.
Cell. Host
Microbe 2012, 11, 607–616.
[Google Scholar]
[CrossRef]
[PubMed]
- Barcena,
M.; Oostergetel, G.T.; Bartelink, W.;
Faas, F.G.; Verkleij, A.; Rottier, P.J.;
Koster, A.J.; Bosch, B.J. Cryo-electron
tomography of mouse hepatitis virus:
Insights into the structure of the
coronavirion. Proc.
Natl. Acad. Sci. USA 2009,
106,
582–587. [Google Scholar]
[CrossRef]
[PubMed]
- Neuman,
B.W.; Adair, B.D.; Yoshioka, C.; Quispe,
J.D.; Orca, G.; Kuhn, P.; Milligan, R.A.;
Yeager, M.; Buchmeier, M.J. Supramolecular
architecture of severe acute respiratory
syndrome coronavirus revealed by electron
cryomicroscopy. J.
Virol. 2006, 80,
7918–7928. [Google Scholar]
[CrossRef]
[PubMed]
- de
Groot, R.J.; Luytjes, W.; Horzinek, M.C.;
van der Zeijst, B.A.; Spaan, W.J.;
Lenstra, J.A. Evidence for a coiled-coil
structure in the spike proteins of
coronaviruses. J.
Mol. Biol. 1987, 196, 963–966.
[Google Scholar]
[CrossRef]
- Stohlman,
S.A.; Baric, R.S.; Nelson, G.N.; Soe,
L.H.; Welter, L.M.; Deans, R.J. Specific
interaction between coronavirus leader RNA
and nucleocapsid protein. J. Virol. 1988,
62,
4288–4295. [Google Scholar]
[CrossRef]
[PubMed]
- Molenkamp,
R.; Spaan, W.J. Identification of a
specific interaction between the
coronavirus mouse hepatitis virus A59
nucleocapsid protein and packaging signal.
Virology
1997, 239,
78–86. [Google Scholar]
[CrossRef]
- Kuo,
L.; Masters, P.S. Functional analysis of
the murine coronavirus genomic RNA
packaging signal. J. Virol. 2013,
87,
5182–5192. [Google Scholar]
[CrossRef]
- Neuman,
B.W.; Kiss, G.; Kunding, A.H.; Bhella, D.;
Baksh, M.F.; Connelly, S.; Droese, B.;
Klaus, J.P.; Makino, S.; Sawicki, S.G.; et
al. A structural analysis of M protein in
coronavirus assembly and morphology. J. Struct. Biol.
2011, 174,
11–22. [Google Scholar]
[CrossRef]
- Armstrong,
J.; Niemann, H.; Smeekens, S.; Rottier,
P.; Warren, G. Sequence and topology of a
model intracellular membrane protein, E1
glycoprotein, from a coronavirus. Nature 1984,
308,
751–752. [Google Scholar]
[CrossRef]
- Godet,
M.; L’Haridon, R.; Vautherot, J.F.; Laude,
H. TGEV corona virus ORF4 encodes a
membrane protein that is incorporated into
virions. Virology
1992, 188,
666–675. [Google Scholar]
[CrossRef]
- Nieto-Torres,
J.L.; Dediego, M.L.; Verdia-Baguena, C.;
Jimenez-Guardeno, J.M.; Regla-Nava, J.A.;
Fernandez-Delgado, R.; Castano-Rodriguez,
C.; Alcaraz, A.; Torres, J.; Aguilella,
V.M.; et al. Severe acute respiratory
syndrome coronavirus envelope protein ion
channel activity promotes virus fitness
and pathogenesis. PLoS Pathogens
2014, 10,
1004077. [Google Scholar]
[CrossRef]
[PubMed]
- Collins,
A.R.; Knobler, R.L.; Powell, H.;
Buchmeier, M.J. Monoclonal antibodies to
murine hepatitis virus-4 (strain JHM)
define the viral glycoprotein responsible
for attachment and cell-cell fusion. Virology 1982,
119,
358–371. [Google Scholar]
[CrossRef]
- Abraham,
S.; Kienzle, T.E.; Lapps, W.; Brian, D.A.
Deduced sequence of the bovine coronavirus
spike protein and identification of the
internal proteolytic cleavage site. Virology 1990,
176,
296–301. [Google Scholar]
[CrossRef]
- Luytjes,
W.; Sturman, L.S.; Bredenbeek, P.J.;
Charite, J.; van der Zeijst, B.A.;
Horzinek, M.C.; Spaan, W.J. Primary
structure of the glycoprotein E2 of
coronavirus MHV-A59 and identification of
the trypsin cleavage site. Virology 1987,
161,
479–487. [Google Scholar]
[CrossRef]
- Walls,
A.C.; Park, Y.J.; Tortorici, M.A.; Wall,
A.; McGuire, A.T.; Veesler, D. Structure,
Function and Antigenicity of the
SARS-CoV-2 Spike Glycoprotein. Cell 2020.
[Google Scholar]
[CrossRef]
[PubMed]
- Klausegger,
A.; Strobl, B.; Regl, G.; Kaser, A.;
Luytjes, W.; Vlasak, R. Identification of
a coronavirus hemagglutinin-esterase with
a substrate specificity different from
those of influenza C virus and bovine
coronavirus. J.
Virol. 1999, 73,
3737–3743. [Google Scholar]
[CrossRef]
[PubMed]
- Cornelissen,
L.A.; Wierda, C.M.; van der Meer, F.J.;
Herrewegh, A.A.; Horzinek, M.C.; Egberink,
H.F.; de Groot, R.J.
Hemagglutinin-esterase, a novel structural
protein of torovirus. J. Virol. 1997,
71,
5277–5286. [Google Scholar]
[CrossRef]
- Kazi,
L.; Lissenberg, A.; Watson, R.; de Groot,
R.J.; Weiss, S.R. Expression of
hemagglutinin esterase protein from
recombinant mouse hepatitis virus enhances
neurovirulence. J.
Virol. 2005, 79,
15064–15073. [Google Scholar]
[CrossRef]
- Kubo,
H.; Yamada, Y.K.; Taguchi, F. Localization
of neutralizing epitopes and the
receptor-binding site within the
amino-terminal 330 amino acids of the
murine coronavirus spike protein. J. Virol. 1994,
68,
5403–5410. [Google Scholar]
[CrossRef]
- Cheng,
P.K.; Wong, D.A.; Tong, L.K.; Ip, S.M.;
Lo, A.C.; Lau, C.S.; Yeung, E.Y.; Lim,
W.W. Viral shedding patterns of
coronavirus in patients with probable
severe acute respiratory syndrome. Lancet 2004,
363,
1699–1700. [Google Scholar]
[CrossRef]
- Nedellec,
P.; Dveksler, G.S.; Daniels, E.; Turbide,
C.; Chow, B.; Basile, A.A.; Holmes, K.V.;
Beauchemin, N. Bgp2, a new member of the
carcinoembryonic antigen-related gene
family, encodes an alternative receptor
for mouse hepatitis viruses. J. Virol. 1994,
68,
4525–4537. [Google Scholar]
[CrossRef]
[PubMed]
- van
Doremalen, N.; Miazgowicz, K.L.;
Milne-Price, S.; Bushmaker, T.; Robertson,
S.; Scott, D.; Kinne, J.; McLellan, J.S.;
Zhu, J.; Munster, V.J. Host species
restriction of Middle East respiratory
syndrome coronavirus through its receptor
dipeptidyl peptidase 4. J. Virol. 2014,
88,
9220–9232. [Google Scholar]
[CrossRef]
[PubMed]
- Li, W.;
Moore, M.J.; Vasilieva, N.; Sui, J.; Wong,
S.K.; Berne, M.A.; Somasundaran, M.;
Sullivan, J.L.; Luzuriaga, K.; Greenough,
T.C.; et al. Angiotensin-converting enzyme
2 is a functional receptor for the SARS
coronavirus. Nature
2003, 426,
450–454. [Google Scholar]
[CrossRef]
[PubMed]
- Bosch,
B.J.; van der Zee, R.; de Haan, C.A.;
Rottier, P.J. The coronavirus spike
protein is a class I virus fusion protein:
Structural and functional characterization
of the fusion core complex. J. Virol. 2003,
77,
8801–8811. [Google Scholar]
[CrossRef]
- Belouzard,
S.; Chu, V.C.; Whittaker, G.R. Activation
of the SARS coronavirus spike protein via
sequential proteolytic cleavage at two
distinct sites. Proc.
Natl. Acad. Sci. USA 2009,
106,
5871–5876. [Google Scholar]
[CrossRef]
- Baranov,
P.V.; Henderson, C.M.; Anderson, C.B.;
Gesteland, R.F.; Atkins, J.F.; Howard,
M.T. Programmed ribosomal frameshifting in
decoding the SARS-CoV genome. Virology 2005,
332,
498–510. [Google Scholar]
[CrossRef]
- Brierley,
I.; Digard, P.; Inglis, S.C.
Characterization of an efficient
coronavirus ribosomal frameshifting
signal: Requirement for an RNA pseudoknot.
Cell 1989,
57,
537–547. [Google Scholar]
[CrossRef]
- Brown,
C.G.; Nixon, K.S.; Senanayake, S.D.;
Brian, D.A. An RNA stem-loop within the
bovine coronavirus nsp1 coding region is a
cis-acting element in defective
interfering RNA replication. J. Virol. 2007,
81,
7716–7724. [Google Scholar]
[CrossRef]
- Guan,
B.J.; Wu, H.Y.; Brian, D.A. An optimal
cis-replication stem-loop IV in the 5’
untranslated region of the mouse
coronavirus genome extends 16 nucleotides
into open reading frame 1. J. Virol. 2011,
85,
5593–5605. [Google Scholar]
[CrossRef]
- Liu,
P.; Li, L.; Keane, S.C.; Yang, D.;
Leibowitz, J.L.; Giedroc, D.P. Mouse
hepatitis virus stem-loop 2 adopts a Uynmg
(U)a-like tetraloop structure that is
highly functionally tolerant of base
substitutions. J.
Virol. 2009, 83,
12084–12093. [Google Scholar]
[CrossRef]
- Madhugiri,
R.; Fricke, M.; Marz, M.; Ziebuhr, J.
Coronavirus cis-Acting RNA Elements. Adv. Virus Res.
2016, 96,
127–163. [Google Scholar]
[PubMed]
- Raman,
S.; Bouma, P.; Williams, G.D.; Brian, D.A.
Stem-loop III in the 5’ untranslated
region is a cisacting element in bovine
coronavirus defective interfering RNA
replication. J.
Virol. 2003, 77,
6720–6730. [Google Scholar]
[CrossRef]
[PubMed]
- Sethna,
P.B.; Hofmann, M.A.; Brian, D.A.
Minus-strand copies of replicating
coronavirus mRNAs contain antileaders. J. Virol. 1991,
65,
320–325. [Google Scholar]
[CrossRef]
[PubMed]
- Sawicki,
S.G.; Sawicki, D.L.; Siddell, S.G. A
contemporary view of coronavirus
transcription. J.
Virol. 2007, 81, 20–29. [Google Scholar]
[CrossRef]
[PubMed]
- Krijnse-Locker,
J.; Ericsson, M.; Rottier, P.J.M.;
Griffiths, G. Characterization of the
budding compartment of mouse hepatitis
virus: Evidence that transport from the
RER to the golgi complex requires only one
vesicular transport step. J. Cell Biol.
1994, 124,
55–70. [Google Scholar]
[CrossRef]
[PubMed]
- Tooze,
J.; Tooze, S.; Warren, G. Replication of
coronavirus MHV-A59 in sac- cells:
Determination of the first site of budding
of progeny virions. Eur. J. Cell Biol.
1984, 33,
281–293. [Google Scholar]
- de
Haan, C.A.; Rottier, P.J. Molecular
interactions in the assembly of
coronaviruses. Adv.
Virus Res. 2005, 64, 165–230.
[Google Scholar]
- Lu, H.
Drug treatment options for the 2019-new
coronavirus (2019.nCoV). Biosci. Trends.
2020. [Google Scholar]
[CrossRef]
- Kruse,
R.L. Therapeutic strategies in an outbreak
scenario to treat the novel coronavirus
originating in Wuhan, China. F1000Res 2020,
9, 72. [Google Scholar]
[CrossRef]
- Wu,
C.J.; Chan, Y.L. Antiviral applications of
RNAi for coronavirus. Exp. Opin. Investig.
Drugs 2006, 15, 89–97. [Google Scholar]
[CrossRef]
- Enjuanes,
L.; Zuniga, S.; Castano-Rodriguez, C.;
Gutierrez-Alvarez, J.; Canton, J.; Sola,
I. Molecular Basis of Coronavirus
Virulence and Vaccine Development. Adv. Virus Res.
2016, 95,
245–286. [Google Scholar]
- Croxtall,
J.D.; Perry, C.M. Lopinavir/Ritonavir: A
review of its use in the management of
HIV-1 infection. Drugs 2010,
70,
1885–1915. [Google Scholar]
[CrossRef]
[PubMed]
- Chan,
J.F.; Yao, Y.; Yeung, M.L.; Deng, W.; Bao,
L.; Jia, L.; Li, F.; Xiao, C.; Gao, H.;
Yu, P.; et al. Treatment with
Lopinavir/Ritonavir or Interferon-β1b
Improves Outcome of MERS-CoV Infection in
a Nonhuman Primate Model of Common
Marmoset. J.
Infect. Dis. 2015, 212,
1904–1913. [Google Scholar]
[CrossRef]
[PubMed]
- Arabi,
Y.M.; Alothman, A.; Balkhy, H.H.;
Al-Dawood, A.; Al Johani, S.; Al Harbi,
S.; Kojan, S.; Al Jeraisy, M.; Deeb, A.M.;
Assiri, A.M. Treatment of Middle East
Respiratory Syndrome with a combination of
lopinavir-ritonavir and interferon-β1b
(MIRACLE) trial: Study protocol for a
randomized controlled trial. Trials 2018,
19, 81. [Google Scholar]
[CrossRef]
- Cao,
B.; Wang, Y.; Wen, D.; Liu, W.; Wang, J.;
Fan, G.; Ruang, L.; Song, B.; Cai, Y.;
Wei, M.; et al. A trial of
Lopinavir-Ritonavir in Adults Hospitalized
with Severe Covid-19. N. Engl. J. Med.
2020. [Google Scholar]
[CrossRef]
- Prujisssers,
A.J.; Denison, M.R. Nucleoside analogues
for the treatment of coronavirus
infections. Curr.
Opin. Virol. 2019, 35, 57–62. [Google Scholar]
[CrossRef]
- Chu,
C.K.; Gadthula, S.; Chen, X.; Choo, H.;
Olgen, S.; Barnard, D.L.; Sidwell, R.W.
Antiviral activity of nucleoside analogues
against SARS-coronavirus (SARS-CoV). Antivir. Chem.
Chemother. 2006, 17, 285–289.
[Google Scholar]
[CrossRef]
- Peters,
H.L.; Jochmans, D.; de Wilde, A.H.;
Posthuma, C.C.; Snijder, E.J.; Neyts, J.;
Seley-Radtke, K.L. Design, synthesis and
evaluation of a series of acyclic fleximer
nucleoside analogues with anti-coronavirus
activity. Bioorg.
Med. Chem. Lett. 2015, 25,
2923–2926. [Google Scholar]
[CrossRef]
- Murphy,
B.G.; Perron, M.; Murakami, E.; Bauer, K.;
Park, Y.; Eckstrand, C.; Liepnieks, M.;
Pedersen, N.C. The nucleoside analog
GS-441524 strongly inhibits feline
infectious peritonitis (FIP) virus in
tissue culture and in experimental cat
infection studies. Vet. Microbiol.
2018, 219,
226–233. [Google Scholar]
[CrossRef]
- Omrani,
A.S.; Saad, M.M.; Baig, K.; Bahloul, A.;
Abdul-Matin, M.; Alaidaroos, A.Y.;
Almakhlafi, G.A.; Albarrak, M.M.; Memish,
Z.A.; Albarrak, A.M. Ribavirin and
interferon alpha-2a for severe Middle East
respiratory syndrome coronavirus
infection: A retrospective cohort study. Lancet Infect. Dis.
2014, 14,
1090–1095. [Google Scholar]
[CrossRef]
- Lo,
M.K.; Jordan, R.; Arvey, A.; Sudhamsu, J.;
Shrivastava-Ranjan, P.; Hotard, A.L.;
Flint, M.; McMullan, L.K.; Siegel, D.;
Clarke, M.O.; et al. GS-5734 and its
parent nucleoside analog inhibit Filo-,
Pneumo and Paramyxoviruses. Sci. Rep. 2017,
7, 43395.
[Google Scholar]
[CrossRef]
[PubMed]
- Brown,
A.J.; Won, J.J.; Graham, R.L.; Dinnon,
K.H., 3rd; Sims, A.C.; Feng, J.Y.; Cihlar,
T.; Denison, M.R.; Baric, R.S.; Sheahan,
T.P. Broad spectrum antiviral remdesivir
inhibits human endemic and zoonotic
deltacoronaviruses with a highly divergent
RNA dependent RNA polymerase. Antiviral Res.
2019, 169,
104541. [Google Scholar]
[CrossRef]
[PubMed]
- Gordon,
C.J.; Tchesnokov, E.P.; Feng, J.Y.;
Porter, D.P.; Gotte, M. The antiviral
compound remdesivir potently inhibits
RNA-dependent RNA polymerase from Middle
East respiratory syndrome coronavirus. J. Biol. Chem.
2020. [Google Scholar]
[CrossRef]
[PubMed]
- Wang,
M.; Cao, R.; Zhang, L.; Yang, X.; Liu, J.;
Xu, M.; Shi, Z.; Hu, Z.; Zhong, W.; Xiao,
G. Remdesivir and chloroquine effectively
inhibit the recently emerged novel
coronavirus (2019-nCoV) in vitro. Cell Res. 2020,
30,
269–271. [Google Scholar]
[CrossRef]
[PubMed]
- Ko,
W.-C.; Rolain, J.-M.; Lee, N.-Y.; Chen,
P.L.; Huang, C.-T.; Lee, P.-I.; Hsueh,
P.-R. Arguments in favour of remdesivir
for treating SARS-CoV-2 infections. Int. J. Antimicrob.
Agents 2020. [Google Scholar]
[CrossRef]
[PubMed]
- Laborda,
P.; Wang, S.Y.; Voglmeir, J. Influenza
Neuraminidase Inhibitors: Synthetic
Approaches, Derivatives and Biological
Activity. Molecules
2016, 21,
1513. [Google Scholar]
[CrossRef]
- Tan,
E.L.C.; Eong Ooi, E.; Lin, C.-Y.; Cheng
Tan, H.; Ling, A.E.; Lim, B.; Stanton,
L.W. Inhibition of SARS Coronavirus
Infection In Vitro with Clinically
Approved Drugs. Emerg.
Inf. Dis. 2004, 10, 581–586.
[Google Scholar]
[CrossRef]
- Haviemik,
J.; Stefanik, M.; Foitikova, M.; Kali, S.;
Tordo, N.; Rudolf, I.; Hubalek, Z.; Eyer,
L.; Ruzek, D. Arbidol (Umifenovir): A
Broad-Spectrum Antiviral Drug That
Inhibits Medically Important
Arthropod-Borne Flaviviruses. Viruses 2018,
10, 184.
[Google Scholar]
[CrossRef]
- Fink,
S.L.; Voitech, L.; Wagoner, J.; Slivinski,
N.S.J.; Jackson, K.J.; Wang, R.; Khadka,
S.; Luthra, P.; Basler, C.F.; Polyak, S.J.
The Antiviral Drug Arbidol Inhibits Zika
Virus. Sci.
Rep. 2018, 8, 8989. [Google Scholar]
[CrossRef]
- Deng,
L.; Li, C.; Zeng, Q.; Liu, X.; Li, X.;
Zhang, H.; Hong, Z.; Xia, J. Arbidol
combined with LPV/r versus LPV/r alone
against Corona Virus Disease 2019: A
retrospective cohort study. J. Infect. 2020.
[Google Scholar]
[CrossRef]
- Gautret,
P.; Lagier, J.-C.; Parola, P.; Hoang,
V.T.; Meddeb, L.; Mailhe, M.; Doudier, B.;
Courjon, J.; Giordanengo, V.; Vieira,
E.V.; et al. Hydroxychloroquine and
azothromycin as a treatment of COVID-19:
Results of an open-label non-randomized
clinical trial. Int.
J. Antimicrob. Agents 2020.
[Google Scholar]
[CrossRef]
[PubMed]
- Ni,
L.; Zhou, L.; Zhou, M.; Zhao, J.; Wang,
D.W. Combination of western medicine and
Chinese traditional patent medicine in
treating a family case of COVID-19 in
Wuhan. Front.
Med. 2020. [Google Scholar]
[CrossRef]
[PubMed]
- Wang,
Z.; Chen, X.; Lu, Y.; Chen, F.; Zhang, W.
Clinical characteristics and therapeutic
procedure for four cases with 2019 novel
coronavirus pneumonia receiving combined
Chinese and Western medicine treatment. Biosci. Trends
2020, 14,
64–68. [Google Scholar]
[CrossRef]
[PubMed]
- Hoffmann,
M.; Kleine-Weber, H.; Schroeder, S.;
Krüger, N.; Herrler, T.; Erichsen, S.;
Schiergens, T.S.; Herrler, G.; Wu, N.;
Nitsche, A.; et al. SARS-CoV-2 Cell Entry
Depends on ACE2 and TMPRSS2 and Is Blocked
by a Clinically Proven Protease Inhibitor.
Cell 2020.
[Google Scholar]
[CrossRef]
[PubMed]
- Tian,
X.; Li, C.; Huang, A.; Xia, S.; Lu, S.;
Shi, Z.; Lu, L.; Jiang, S.; Yang, Z.; Wu,
Y.; et al. Potent binding of novel 2019
coronavirus spike protein by a SARS
coronavirus-specific human monoclonal
antibody. Emerg.
Mircobes. Infect. 2020, 9, 382–385. [Google Scholar]
[CrossRef]
[PubMed]
- Gurwitz,
D. Angiotensin receptor blockers as
tentative SARS-CoV-2 therapeutics. Drug Dev. Res.
2020. [Google Scholar]
[CrossRef]
- Wang,
C.; Li, W.; Drabek, D.; Okba, N.M.A.; van
Haperen, R.; Osterhaus, A.D.; van
Kupperveld, F.; Haagmans, B.L.; Grosveld,
F.; Bosch, B.-J. A human monoclonal
antibody blocking SARS-CoV infection. bioRxiv 2020.
preprint. [Google Scholar]
[CrossRef]
- Vincent,
M.J.; Bergeron, E.; Benjannet, S.;
Erickson, B.R.; Rollin, P.E.; Ksiazek,
T.G.; Seidah, G.S.; Nichol, S.T.
Chlkoroquine is a potent inhibitor of SARS
coronavirus infection and spread. Virol. J. 2005,
2, 69. [Google Scholar]
[CrossRef]
- Colson,
P.; Rolain, J.-M.; Raoult, D. Chloroquine
for the 2019 novel coronavirus SARS-CoV-2.
Int. J.
Antimicrob. Agents 2020,
55,
105923. [Google Scholar]
[CrossRef]
- Zhou,
D.; Dai, S.M.; Tong, Q. COVID-19: A
recommendation to examine the effect of
hydrochloroquine in preventing infection
and progression. J. Antimicrob.
Chemother. 2020, 114. [Google Scholar]
[CrossRef]
- Di
Pierro, F.; Bertuccioli, A.; Cavecchia, I.
Possible therapeutic role of a highly
standardized mixture of active compounds
derived from cultured Lentinula edodes
mycelia (AHCC) in patients infected with
2019 novel coronavirus. Minerva.
Gastoenterol. Dietol. 2020.
[Google Scholar]
[CrossRef]
- Ding,
Y.; Zeng, L.; Li, R.; Chen, Q.; Zhou, B.;
Chen, Q.; Cheng, P.L.; Yutao, W.; Zheng,
J.; Yang, Z. The Chinese prescription
lianhuaqingwen capsule exerts
anti-influenza activity through the
inhibition of viral propagation and
impacts immune function. BMC Complement
Altern. Med. 2017, 17, 130. [Google Scholar]
[CrossRef]
[PubMed]
- Moen,
M.D.; Wagstaff, A.J. Losartan: A review of
its use in stroke risk reduction in
patients with hypertension and left
ventricular hypertrophy. Drugs 2005,
65,
2657–2674. [Google Scholar]
[CrossRef]
[PubMed]
- Lee,
R.C.; Feinbaum, R.L.; Ambros, V.; The, C.
elegans heterochronic gene lin-4 encodes
small RNAs with antisense complimentary to
lin-14. Cell
1993, 75,
843–854. [Google Scholar]
[CrossRef]
- Lundstrom,
K. Micro-RNA in disease and gene therapy.
Curr. Drug
Discov. Technol. 2011, 8, 76–86. [Google Scholar]
[CrossRef]
- Shi,
Y. Mammalian RNAi for the masses. Trends Genet.
2003, 19,
9–12. [Google Scholar]
[CrossRef]
- Zhang,
Y.; Li, T.; Fu, L.; Yu, C.; Li, Y.; Xu,
X.; Wang, Y.; Ning, H.; Zhang, S.; Chen,
W.; et al. Silencing SARS-CoV Spike
protein expression in cultured cells by
RNA interference. FEBS Lett. 2004,
560,
141–146. [Google Scholar]
[CrossRef]
- Zheng,
B.J.; Guan, Y.; Tang, Q.; Du, C.; Xie,
F.Y.; He, M.L.; Chan, K.W.; Wong, K.L.;
Lader, E.; Woodle, M.C.; et al.
Prophylactic and therapeutic effects of
small interfering RNA targeting
SARS-coronavirus. Antivir. Res.
2004, 9,
365–374. [Google Scholar]
- Chang,
Z.; Hu, J. Can siRNA conquer SARS? Gene Ther. 2006,
13,
871–872. [Google Scholar]
[CrossRef]
- Lu,
C.Y.; Huang, H.-Y.; Yang, T.H.; Chang,
L.Y.; Lee, C.Y.; Huang, L.M. siRNA
silencing of angiotensin-converting enzyme
2 reduced severe acute respiratory
syndrome-associated coronavirus
replications in Vero cells. Eur. J. Clin.
Microbiol. Infect. Dis. 2008,
27,
709–715. [Google Scholar]
[CrossRef]
- Millet,
J.K.; Nal, B. Investigation of the
functional roles of host cell proteins
involved in coronavirus infection using
highly specific and scalable RNA
interference (RNAi) approach. Methods Mol. Biol.
2015, 1282,
231–240. [Google Scholar]
[PubMed]
- Nur,
S.M.; Hasan, M.A.; Amin, M.A.; Hossain,
M.; Sharmin, T. Design of Potential RNAi
(miRNA and siRNA) Molecules for Middle
East Respiratory Syndrome Coronavirus
(MERS-CoV) Gene Silencing by Computational
Method. Interdiscip.
Sci. 2015, 7, 257–265. [Google Scholar]
[CrossRef]
[PubMed]
- McDonagh,
P.; Sheehy, P.A.; Norris, J.M. Combination
siRNA therapy against feline coronavirus
can delay the emergence of antiviral
resistance in vitro. Vet. Microbiol.
2015, 176,
10–18. [Google Scholar]
[CrossRef]
[PubMed]
- Gu,
W.Y.; Li, Y.; Liu, B.J.; Wang, J.; Yuan,
G.F.; Chen, S.J.; Zuo, Y.Z.; Fan, J.H.
Short hairpin RNAs targeting M and N genes
reduce replication of porcine
deltacoronavirus in ST cells. Virus Genes 2019,
55,
795–801. [Google Scholar]
[CrossRef]
- Li,
K.; Li, H.; Bi, Z.; Song, D.; Zhang, F.;
Lei, D.; Luo, S.; Li, Z.; Gong, W.; Huang,
D.; et al. Significant inhibition of
re-emerged and emerging swine enteric
coronavirus in vitro using the multiple
shRNA expression vector. Antiviral. Res.
2019, 166,
11–18. [Google Scholar]
[CrossRef]
- Sohrab,
S.S.; El-Kafrawy, S.A.; Mirza, Z.; Kamal,
M.A.; Azhar, E.L. Design and Delivery of
Therapeutic siRNAs: Application to
MERS-Coronavirus. Curr. Pharm. Des.
2018, 24,
62–77. [Google Scholar]
[CrossRef]
- Delrue,
I.; Verzele, D.; Madder, A.; Nauwynck,
H.J. Inactivated virus vaccines: From
chemistry to prophylaxis: Merits, risks
and challenges. Expert Rev. Vaccines 2012,
11,
695–719. [Google Scholar]
[CrossRef]
- Deng,
M.P.; Hu, Z.H.; Wang, H.L.; Deng, F.
Developments of subunit and VLP vaccines
against influenza A virus. Virol. Sin. 2012,
27,
145–153. [Google Scholar]
[CrossRef]
- Decaro,
N.; Campolo, M.; Mari, V.; Desario, C.;
Colaianni, M.L.; Di Trani, L.; Cordioli,
P.; Buonavoglia, C. A candidate
modified-live bovine coronavirus vaccine:
Safety and immunogenicity evaluation. New Microbiol.
2009, 32,
109–132. [Google Scholar]
- Bovilis®
Coronavirus. Available online:
https://www.merck-animal-health-usa.com/product/
cattle/Bovilis-Coronavirus/1
(accessed on 25 March 2020).
- Nemoto,
M.; Kanno, T.; Bannai, H.; Tsujimura, K.;
Yamanaka, T.; Kokado, H. Antibody response
to equine coronavirus in horses inoculated
with a bovine coronavirus vaccine. Vet. Med. Sci.
2017, 79,
1889–1891. [Google Scholar]
[CrossRef]
- Ibrahim,
H.S.; Kafi, S.K. A Computational Vaccine
Design Approach for MESR-CoV Infections. Methods Mol. Biol.
2020, 2131,
39–145. [Google Scholar]
[PubMed]
- Battacharaya,
M.; Sharma, A.R.; Patra, P.; Ghosh, P.;
Sharma, G.; Patra, B.C.; Lee, S.S.;
Chakraborty, C. Development of
epitope-based peptide vaccine against
novel coronavirus 2019 (SARS-CoV-2):
Immunoinformatics approach. J. Med. Virol.
2020. [Google Scholar]
[CrossRef]
[PubMed]
- Tahir,
U.I.; Qamar, M.; Saleem, S.; Ashfaq, U.A.;
Bari, A.; Anwar, F.; Alqatani, S.
Epitope-based peptide vaccine design and
target site depiction against Middle East
Respiratory Syndrome Coronavirus: An
immune-informatics study. J. Transl. Med.
2019, 17,
362. [Google Scholar]
[CrossRef]
- Srivastava,
S.; Kamthania, M.; Singh, S.; Saxena,
A.K.; Sharma, N. Structural basis of
development of multi-epitope vaccine
against Middle East respiratory syndrome
using in silico approach. Infect. Drug Resist.
2018, 11,
2377–2391. [Google Scholar]
[CrossRef]
[PubMed]
- Wang,
N.; Rosen, O.; Wang, L.; Turner, H.L.;
Stevens, L.J.; Corbett, K.S.; Bowman,
C.A.; Pallesen, J.; Shi, W.; Zhang, Y.; et
al. Structural Definition of a
Neutralization-Sensitive Epitope on the
MERS-CoV S1-NTD. Cell Rep. 2019,
28,
3395–3405. [Google Scholar]
[CrossRef]
[PubMed]
- Walls,
A.C.; Tortorici, M.A.; Frenz, B.; Snjider,
J.; Li, W.; Rey, F.A.; DiMaio, F.; Bosch,
B.J.; Veesler, D. Glycan shield and
epitope masking of a coronavirus spike
protein observed by cryo-electron
microscopy. Nat.
Struct. Mol. Biol. 2016,
23,
899–905. [Google Scholar]
[CrossRef]
- Ahmed,
S.F.; Quadeer, A.A.; McKay, M.R.
Preliminary Identification of Potential
Vaccine Targets for the COVID-19
Coronavirus (SARS-CoV-2) Based on SARS-CoV
Immunological Studies. Viruses 2020,
12, 254.
[Google Scholar]
[CrossRef]
- Zakhartchouk,
A.N.; Viswanathan, N.; Moshynskyy, I.;
Petric, M.; Babiuk, L.A. Optimization of a
DNA vaccine against SARS. DNA Cell Biol.
2007, 26,
721–726. [Google Scholar]
[CrossRef]
- Callendret,
B.; Lorin, V.; Charneau, P.; Marianneau,
P.; Contamin, H.; Betton, J.M.; van der
Werf, S.; Escriou, N. Heterologous viral
RNA export elements improve expression of
severe acute respiratory syndrome (SARS)
coronavirus spike protein and protective
efficacy of DNA vaccines against SARS. Virology 2007,
363,
288–302. [Google Scholar]
[CrossRef]
- Shim,
B.S.; Park, S.M.; Quan, J.S.; Jere, D.;
Chu, H.; Song, M.K.; Kim, D.W.; Jang,
Y.S.; Yang, M.S.; Han, S.H.; et al.
Intranasal immunization with plasmid DNA
encoding spike protein of
SARS-coronavirus/polyethylenimine
nanoparticles elicits antigen-specific
humoral and cellular immune responses. BMC Immunol.
2010, 11,
65. [Google Scholar]
[CrossRef]
- Chen,
Y.N.; Wu, C.C.; Yeo, Y.; Xu, P.; Lin, T.L.
A DNA prime-protein boost vaccination
strategy targeting turkey coronavirus
spike protein fragment containing
neutralizing epitope against infectious
challenge. Vet.
Immunol. Immunopathol. 2013,
152,
359–369. [Google Scholar]
[CrossRef]
[PubMed]
- Xue,
R.; Tian, Y.; Zhang, Y.; Zhang, M.; Tian,
F.; Ma, J.; Jiang, S. Efficacy and
immunogenicity of a live L. acidophilus
expressing SAD epitope of transmissible
gastroenteritis virus as an oral vaccine.
Acta Virol.
2019, 63,
301–308. [Google Scholar]
[CrossRef]
[PubMed]
- Nyon,
M.P.; Du, L.; Tseng, C.K.; Seid, C.A.;
Pollet, J.; Naceanceno, K.S.; Agrawal, A.;
Algaissi, A.; Peng, B.H.; Tai, W.; et al.
Engineering a stable CHO cell line for the
expression of a MERS-coronavirus vaccine
antigen. Vaccine
2018, 36,
1853–1862. [Google Scholar]
[CrossRef]
[PubMed]
- Li,
H.Y.; Ramalingam, S.; Chye, M.L.
Accumulation of recombinant SARS-CoV spike
protein in cytosol and chloroplasts
indicate potential for development of
plant-derived oral vaccines. Exp. Biol. Med.
2006, 231,
1346–1352. [Google Scholar]
[CrossRef]
- Pogrebnyak,
N.; Golovkin, M.; Andrianov, V.; Spitsin,
S.; Smirnov, Y.; Egolf, R.; Koprowski, H.
Severe acute respiratory syndrome (SARS) S
protein production in plants: Development
of recombinant vaccine. Proc. Natl. Acad.
Sci. USA 2005, 102,
9062–9067. [Google Scholar]
[CrossRef]
- Zheng,
N.; Xia, R.; Yang, C.; Yin, B.; Li, Y.;
Duan, C.; Liang, L.; Guo, H.; Xie, Q.
Boosted expression of the SARS-CoV
nucleocapsid protein in tobacco and its
immunogenicity in mice. Vaccine 2009,
27,
5001–5007. [Google Scholar]
[CrossRef]
- Do,
V.T.; Jang, J.; Park, J.; Dao, H.T.; Kim,
K.; Hahn, T.W. Recombinant adenovirus
carrying a core neutralizing epitope of
porcine epidemic diarrhea virus and
heat-labile enterotoxin B of Escherichia
coli as a mucosal vaccine. Arch. Virol.
2020, 165,
609–618. [Google Scholar]
[CrossRef]
- Haagmans,
B.L.; van den Brand, J.M.; Raj, V.S.;
Volz, A.; Wohlsein, P.; Smits, S.L.;
Schipper, D.; Bestebroer, T.M.; Okba, N.;
Fux, R.; et al. An orthopoxvirus-based
vaccine reduces virus excretion after
MERS-CoV infection in dromedary camels. Science 2016,
351,
77–81. [Google Scholar]
[CrossRef]
- Veit,
S.; Jany, S.; Fux, R.; Sutter, G.; Volz,
A. CD8+ T Cells Responding to the Middle
East Respiratory Syndrome Coronavirus
Nucleocapsid Delivered by Vaccinia Virus
MVA in Mice. Viruses
2018, 10,
718. [Google Scholar]
[CrossRef]
- Faber,
M.; Lamirande, E.W.; Roberts, A.; Rice,
A.B.; Koprowski, H.; Dietschold, B.;
Schnell, M.J. A single imunization with a
rhabdovirus-based vector expressing severe
acute respiratory syndrome coronavirus
(SARS-CoV) S protein results in the
production of high levels of
SARS-CoV-neutralizing antibodies. J. Gen. Virol.
2005, 86,
1435–1440. [Google Scholar]
[CrossRef]
- Deming,
D.; Sheahan, T.; Heise, M.; Yount, B.;
Davis, N.; Sims, A.; Suthar, M.; Harkema,
J.; Whitmore, A.; Pickles, R.; et al.
Vaccine efficacy in senescent mice
challenged with recombinant SARS-CoV
bearing epidemic and zoonotic spike
variants. PLoS
Med. 2006, 3, e525. [Google Scholar]
[CrossRef]
[PubMed]
- Sheahan,
T.; Whitmore, A.; Long, K.; Ferris, M.;
Rockx, B.; Funkhouser, B.; Donaldson, E.;
Gralinski, L.; Collier, M.; Heise, M.; et
al. Successful vaccination strategies that
protect aged mice from lethal challenge
from influenza virus and heterologous
severe acute respiratory syndrome
coronavirus. J.
Virol. 2011, 85, 217–230.
[Google Scholar]
[CrossRef]
[PubMed]
- Bennett,
R.P.; Dalby, B. Protein delivery using
VP22. Nat.
Biotechnol. 2002, 20, 20. [Google Scholar]
[CrossRef]
[PubMed]
- Almazan,
F.; Galan, C.; Enjuanes, L. Engineering
infectious cDNAs of coronavirus as
bacterial artificial chromosomes. Methods Mol. Biol.
2008, 454,
275–291. [Google Scholar]
[PubMed]
- Shanmugarai,
B.; Malla, A.; Phoolcharoen, W. Emergence
of Novel Coronavirus 2019-nCoV: Need for
Rapid Vaccine and Biologics Development. Pathogens 2020,
9, 148. [Google Scholar]
[CrossRef]
[PubMed]
- Lundstrom,
K. RNA Viruses as Tools in Gene Therapy
and Vaccine Development. Genes 2019,
10, 189.
[Google Scholar]
[CrossRef]
- Lundstrom,
K. Plasmid DNA-based Alphavirus Vaccines.
Vaccines
2019, 7,
29. [Google Scholar]
[CrossRef]
- Francesco,
N. Is antibody-dependent enhancement
playing a role in COVID-19 pathogenesis? Swiss. Med. Wkly.
2020, 150,
w20249. [Google Scholar]
- Tetro,
J.A. Is COVID-19 receiving ADE from other
coronaviruses? Microbes.
Inf. 2020, 22, 72–73. [Google Scholar]
[CrossRef]
- Wan,
Y.; Shang, J.; Sun, S.; Tan, W.; Chen, J.;
Geng, Q.; He, L.; Chen, Y.; Wu, J.; Shi,
Z.; et al. Molecular Mechanism for
Antibody-Dependent Enhancement of
Coronavirus Entry. J. Virol. 2020,
94,
e02015-19. [Google Scholar]
[CrossRef]
- Chen,
K.; Kolls, J.K. T cell-mediated host
immune defenses in the lung. Annu. Rev. Immunol.
2013, 31,
605–633. [Google Scholar]
[CrossRef]
[PubMed]
- Mason,
R.J. Pathogenesis of COVID-19 from a cell
biologic perspective. Eur. Respir. J.
2020, 55,
2000607. [Google Scholar]
[CrossRef]
[PubMed]
- Bloch,
E.M.; Shoham, S.; Casadevall, A.; Sachais,
B.S.; Shaz, B.; Winters, J.L.; van
Burskirk, C.; Grossman, B.J.; Joyner, M.;
Henderson, J.P.; et al. Deployment of
convalescent plasma for the prevention and
treatment of COVID-19. J. Clin. Investig.
2020, 138745. [Google Scholar]
[CrossRef]
[PubMed]
- Zhang,
L.; Pang, R.; Xue, X.; Bao, J.; Ye, S.;
Daj, Y.; Zheng, Y.; Fu, Q.; Hu, Z.; Yi, Y.
Anti-SARS-CoV-2 virus antibody levels in
convalescent plasma of six donors who have
recovered from COVID-19. Aging 2020,
12. [Google Scholar]
[CrossRef]
- Shen,
C.; Wang, Z.; Zhao, F.; Yang, Y.; Li, J.;
Yuan, J.; Wang, F.; Li, D.; Yang, M.;
Xing, L.; et al. Treatment of 5 Critically
Ill Patients with COVID-19 With
Convalescent Plasma. JAMA 2020.
[Google Scholar]
[CrossRef]
- Grein,
J.; Ohmagari, N.; Shin, D.; Diaz, G.;
Asperges, E.; Castagna, A.; Feldt, T.;
Green, G.; Green, M.L.; Lescure, F.X.; et
al. Compassionate Use of Remdesivir for
Patients with COVID-19. N. Engl. J. Med.
2020. [Google Scholar]
[CrossRef]
- Available
online: www.gilead.com/news-and-press/company-statements/gilead-sciences-statement-on-data-from-remdesivir-study-in-patients-with-
severe-covid-19-in-china (accessed
on 30 April 2020).
- Available
online: www.theguardian.com/world/2020/apr/23/high-hopes-drug-for-covid-19-treatment-failed-in-full-trial
(accessed on 30 April 2020).
- Ferner,
R.E.; Aronson, J.K. Chloroquine and
hydroxychloroquine in COVID-19. BMJ 2020,
369,
m1432. [Google Scholar]
[CrossRef]
- Gao,
J.; Tian, Z.; Yang, X. Breakthrough:
Chloroquine phosphate has shown apparent
efficacy in treatment of COVID-19
associated pneumonia in clinical studies.
Biosci. Trends
2020, 14,
72–73. [Google Scholar]
[CrossRef]
- Chen,
Z.; Hu, J.; Zhang, Z.; Jiang, S.; Han, S.;
Yan, D.; Zhuang, R.; Hu, B.; Zhang, Z.
Efficacy of hydroxychloroquine in patients
with COVID-19: Results of a randomized
clinical trial. Version 2. medRxiv 2020.
[Google Scholar]
[CrossRef]
- Yan,
D.; Zhang, Z. Therapeutic Effect of
Hydroxychloroquine on Novel Coronavirus
Pneumonia (COVID-19). Chinese Clinical
Trials Registry. Available online: http://www.chictr.org.cn/
showproj.aspx?proj=48880 (accessed
on 28 April 2020).
- Murphy,
M.; Carmichael, A.J. Fatal toxic epidermal
necrolysis associated with
hydroxychloroquine. Clin. Exp. Dermatol.
2001, 26,
457–458. [Google Scholar]
[CrossRef]
- Makin,
A.J.; Wendon, J.; Fitt, S.; Portmann,
B.C.; Williams, R. Fulminant hepatic
failure secondary to hydroxychloroquine. Gut 1994,
35,
569–570. [Google Scholar]
[CrossRef]
[PubMed]
- Chorin,
E.; Dai, M.; Shulman, E.; Wadhwani, L.;
Bar-Cohen, R.; Barbhayia, C.; Aizer, A.;
Holmes, D.; Bernstein, S.; Spinelli, M.;
et al. The QT interval in patients with
SARS-CoV-2 infection treated with
hydroxychloroquine/azithromycin. medRxiv 2020.
[Google Scholar]
[CrossRef]
- Li,
Y.; Xie, Z.; Lin, W.; Cai, W.; Wen, C.;
Guan, Y.; Mo, X.; Wang, J.; Wang, Y.;
Peng, P.; et al. An exploratory
randomized, controlled study on the
efficacy and safety of lopinavir/ritonavir
or arbidol treating adult patients
hospitalized with mild/moderate COVID-19
(ELACOI). medRxiv
2020. [Google Scholar]
[CrossRef]
- Available
online: www.who.int/blueprint/
priority-diseases/key-action/novel-
coronavirus-landscape-ncov.pdf
(accessed on 28 April 2020).
- Zhao,
H. COVID-19 drives new threats to bats in
China. Science
2020, 367,
1436. [Google Scholar]
- Cyranoski,
D. Mystery deepens over animal source of
coronavirus. Nature
2020, 579,
18–19. [Google Scholar]
[CrossRef]
© 2020 by the author. Licensee MDPI, Basel,
Switzerland. This article is an open access
article distributed under the terms and
conditions of the Creative Commons Attribution
(CC BY) license (http://creativecommons.org/licenses/by/4.0/).
|
Übersetzung:
Coronavirus-Pandemie - Therapie und
Impfstoffe
von Kenneth Lundstrom
PanTherapeutics, CH1095 Lutry, Schweiz
Biomedicines 2020, 8 (5), 109;
https://doi.org/10.3390/biomedicines8050109
Eingegangen am 31. März 2020 / Überarbeitet am 29.
April 2020 / Akzeptiert am 30. April 2020 /
Veröffentlicht am 3. Mai 2020
(Dieser Artikel gehört zur Sonderausgabe Gentherapie
Coming of Age)
PDF herunterladen Zahlen durchsuchen
Berichte überprüfen
Zusammenfassung
Die derzeitige COVID-19-Pandemie des Coronavirus,
die ihren Ursprung in Wuhan, China, hat neben
erheblichen medizinischen Problemen auch erhebliche
soziale, psychologische und wirtschaftliche Bedenken
aufgeworfen. Die rasche Ausbreitung des schweren
akuten respiratorischen Syndroms Coronavirus
(SARS-CoV) -2 in fast allen Ländern der Welt und das
Versäumnis, die Infektionen einzudämmen, haben
weltweit zu Angst und Panik beigetragen. Der Mangel
an verfügbaren und wirksamen antiviralen
Medikamenten oder Impfstoffen hat die Situation
weiter verschlechtert. Aus diesen Gründen kann nicht
genug betont werden, dass beschleunigte
Anstrengungen zur Entwicklung neuartiger
Arzneimittel und Impfstoffe erforderlich sind. In
diesem Zusammenhang sind neue Ansätze sowohl in der
Gentherapie als auch in der Impfstoffentwicklung
unerlässlich. Frühere Erfahrungen aus SARS- und
MERS-Coronavirus-Impfstoff- und
Arzneimittelentwicklungsprojekten haben sich auf
Glykoprotein-Epitope, monoklonale Antikörper,
Angiotensinrezeptorblocker und
Gen-Silencing-Technologien konzentriert, die auch
für COVID-19 nützlich sein können. Darüber hinaus
wurden bestehende antivirale Mittel, die für andere
Arten von Virusinfektionen verwendet werden, als
dringende Maßnahmen angesehen. Diese Übersicht soll
einen Hintergrund über die Genetik und Biologie von
Coronaviren, Beispiele für therapeutische Strategien
und Impfstrategien sowie mögliche innovative neue
Ansätze liefern.
Schlüsselwörter: Coronavirus; Peptid-Impfstoff;
virale Vektoren; Gen-Silencing; RNA-Interferenz;
Virale Replikation
1. Einleitung - [Coronaviren auch bei Tieren]
Coronaviren gehören zur Familie der Coronaviridae
und sind dadurch gekennzeichnet, dass sie
Infektionen der Atemwege verursachen, die von
leichten Krankheiten wie Erkältung bis hin zu
Lungenentzündung mit tödlichem Ausgang reichen [1].
Typischerweise wurden Coronaviren mit einer großen
Anzahl von Krankheiten bei Nutztieren und Haustieren
wie Schweinen, Kühen, Hühnern, Katzen und Hunden in
Verbindung gebracht (Tabelle 1) [2]. In diesem
Zusammenhang sind das übertragbare
Gastroenteritis-Virus (TGEV) [3] und das
Schweine-Epidemie-Durchfall-Virus (PEDV) [4] für
eine signifikante Morbidität und Mortalität bei
jungen Ferkeln verantwortlich. In ähnlicher Weise
verursacht das hämagglutinierende
Schweine-Enzephalomyelitis-Virus (PHEV) bei
Schweinen eine enterische Infektion, kann aber auch
zu einer Enzephalitis führen, indem es auf das
Nervensystem abzielt [5]. Bei Rindern ist Rinder-CoV
(BCoV) für leichte bis schwere Infektionen der
Atemwege verantwortlich, was zu erheblichen
Verlusten in der Rinderindustrie aufgrund von
Durchfall, Dehydration, verminderter Milchproduktion
und Depression führt [6,7]. Neben Rindern infiziert
BCoV auch andere Wiederkäuer wie Elche, Hirsche und
Kamele. Ein anderes Coronavirus, Ratten-CoV (RCoV),
verursacht Atemwegsinfektionen bei Ratten und bietet
ein nützliches Modell für die Untersuchung früher
Ereignisse angeborener Immunantworten auf
Coronavirus-Infektionen in der Lunge [8]. Das
infektiöse Bronchitis-Virus (IBV) zielt auf Hühner
ab und verursacht Infektionen der Atemwege, aber
auch Nierenerkrankungen [9]. IBV hat einen
signifikanten negativen Einfluss auf die
Eiproduktion und das Wachstum von Hühnern, was zu
erheblichen Verlusten in der Hühnerindustrie führt
[7]. Bei Hauskatzen wurde eine milde oder
asymptomatische Infektion mit dem enterischen
Katzen-Coronavirus (FCoV) in Verbindung gebracht
[10], obwohl ein hochvirulenter Stamm des
katzenartigen infektiösen Peritonitis-Virus (FIPV)
eine tödliche infektiöse Katzen-Peritonitis (FIP)
verursacht [11], was zeigt Ähnlichkeiten zur
menschlichen Sarkoidose [12].
Tabelle 1. Coronavirus-basierte Krankheiten
bei Tieren und Menschen.
[Coronavirus bei einem Walfisch]
Interessanterweise können wild lebende Tiere auch
mit Coronaviren infiziert werden. Beispielsweise
wurde aus einem verstorbenen Beluga-Wal ein neues
Coronavirus SW1 isoliert [13].
Der Wal hatte an einer Atemwegserkrankung und einem
akuten Leberversagen gelitten, und große Mengen von
SW1-Partikeln wurden aus der Leber isoliert. Die
Elektronenmikroskopie konnte das Virus jedoch nicht
als Coronavirus identifizieren, aber die
Nukleinsäuresequenzierung von Lebergewebe bestätigte
das Vorhandensein von Coronavirus-RNA. Die
phylogenetische Analyse zeigte, dass SW1 zur Gruppe
der γ-Coronaviren gehörte.
[Coronavirus19 und Fledermäuse]
Aus offensichtlichen Gründen bestand ein starkes
Interesse an Fledermaus-Coronaviren, da Fledermäuse
als ursprüngliche Quelle mehrerer
Coronavirus-Ausbrüche angegeben wurden. In den
letzten zehn Jahren wurden Hunderte neuartiger
Fledermaus-Coronaviren identifiziert [14]. In diesem
Zusammenhang wurde vorgeschlagen, dass sowohl das
schwere akute respiratorische Syndrom-Coronavirus
(SARS-CoV) als auch das nahöstliche respiratorische
Syndrom-CoV (MERS-CoV) von Fledermäusen stammen.
Darüber hinaus wurde bereits 2015 diskutiert, dass
ein SARS-ähnlicher Cluster von
Fledermaus-Coronaviren eine Bedrohung für
menschliche Epidemien darstellen könnte [15].
Beispielsweise wurde das Krankheitspotential eines
SARS-ähnlichen Virus, SHC014-CoV, das in
chinesischen Hufeisenfledermäusen zirkuliert, durch
Umkehrgenetik bewertet [15]. Das chimäre Virus, das
aus SHC014-CoV-Spikes und Wildtyp-Rückgrat besteht,
konnte mehrere Orthologe des SARS-Rezeptors, des
humanen Angiotensin-Converting-Enzyms 2 (ACE2),
effizient nutzen, sich in primären menschlichen
Atemwegszellen auf hohe Werte replizieren und
In-vitro-Titer erzeugen äquivalent zu epidemischen
SARS-CoV-Stämmen. Darüber hinaus zeigte das chimäre
Virus eine Replikation in der Mauslunge mit einer
bemerkenswerten Pathogenese in vivo. Diese
Ergebnisse zeigten, dass ein potenzielles Risiko für
das Wiederauftreten von SARS-CoV durch in
Fledermauspopulationen zirkulierende Viren besteht.
Es ist unnötig zu erwähnen, dass das Wiederauftreten
von SARS-CoV heute Realität ist, wie weiter unten
erläutert wird.
Das murine Hepatitis-Virus (MHV) ist das am besten
untersuchte tierische Coronavirus, das zu
Infektionen der Atemwege, des Darms, der Leber und
der Neurologie führt, und ist auch ein nützliches
Krankheitsmodell [16]. Beispielsweise verursacht
MHV-1 bei A / J- und C3H / HeI-Mäusen schwere
Infektionen der Atemwege. A59 und MHV-3 sind mit
schwerer Hepatitis assoziiert, und JHMV verursacht
schwere Enzephalitis. Interessanterweise verursachen
A59 und ein abgeschwächtes JHMV bei Mäusen eine
chronische demyelinisierende Erkrankung, die der
Multiplen Sklerose (MS) ähnelt und ein Mausmodell
für MS darstellt [17]. Ein weiterer Vorteil von MHV
ist das Erfordernis von Laborbedingungen auf
BSL2-Ebene, während SARS-CoV und MERS-CoV
Laborbedingungen auf BSL3-Ebene erfordern, was MHV
ideal für Studien zur Replikation in Zellkulturen
sowie zur Pathogenese und Immunantwort in vivo
macht.
Im Zusammenhang mit humanen Coronaviren wurde
angenommen, dass sie bis zum SARS-CoV-Ausbruch in
den Jahren 2002–2003 nur leichte selbstlimitierende
Infektionen verursachten [2]. Zwei humane
α-Coronaviren (HCoV-229E und HCoV-NL63) und zwei
β-Coronaviren (HCoV-OC43 und HCoV-HKU1) wurden in
menschlichen Populationen als endemisch
identifiziert und sind für 15 bis 30% der jährlichen
Infektionen der Atemwege verantwortlich [18 20]. Bei
Neugeborenen, älteren Menschen und Personen mit
bereits bestehenden Krankheiten wurde jedoch eine
schwerere Erkrankung festgestellt. Darüber hinaus
wurde HCoV-NL63 auch mit akuter Laryngotracheitis in
Verbindung gebracht [19]. Interessanterweise
unterscheiden sich humane Coronaviren in ihrer
genetischen Variabilität, da aus der ganzen Welt
isoliertes HCoV-229E nur eine minimale
Sequenzdivergenz zeigte [23,24,25,26,27,28,29,30],
während HCoV-OC43 an derselben Stelle isoliert wurde
in verschiedenen Jahren zeigten signifikante
genetische Variabilität [31]. Aus diesem Grund
überschreitet HCoV-229E keine Artenbarrieren und
kann keine Mäuse infizieren, während Mäuse und
mehrere Wiederkäuer anfällig für HCoV-OC43 und das
eng verwandte BCoV sind. Wie oben erwähnt, gibt es,
obwohl MHV die Fähigkeit besitzt, eine
demyelinisierende Krankheit zu verursachen, keine
Hinweise darauf, dass humane Coronaviren mit MS
assoziiert sind.
[Coronavirus SARS 2002-2003]
Der erste große Ausbruch des menschlichen
Coronavirus, der durch das β-Coronavirus SARS-CoV
verursacht wurde, hatte seinen Ursprung in Guangdong
in China [21]. Während der Epidemie 2002–2003 wurden
ungefähr 8098 Fälle registriert, mit 774 Todesfällen
bei einer Sterblichkeitsrate von 9% [22]. Die
Sterblichkeitsrate war jedoch bei älteren Menschen
viel höher und erreichte bei Personen über 60 Jahren
fast 50%. Obwohl eng verwandte Viren aus exotischen
Tieren wie Himalaya-Palmenzibeten und Waschbärhunden
isoliert wurden [24], stammte SARS-CoV auf der
Grundlage von Sequenzdaten und serologischen
Beweisen aus chinesischen Hufeisenfledermäusen [25,
26].
Im Zusammenhang mit SARS-CoV wurden 2013 zwei
neuartige Fledermaus-SARS-bezogene CoVs
identifiziert, die SARS-CoV ähnlicher sind als jedes
andere Virus [27]. Sie verwenden denselben Rezeptor,
ACE2, wie SARS-CoV, ein weiterer Hinweis darauf,
dass SARS-CoV von Fledermäusen stammt. Der
SARS-Ausbruch wurde nur durch direkten Kontakt mit
infizierten Personen aufgrund der relativ
ineffizienten Übertragung des Virus verbreitet,
wodurch der Ausbruch in Haushalten und
Gesundheitseinrichtungen eingedämmt werden konnte
[28]. Daher war der SARS-CoV-Ausbruch durch
Quarantäne kontrollierbar und starb im Juni 2003
aus.
Nach der SARS-CoV-Epidemie trat 2012 im Nahen Osten
das neuartige humane MERS-CoV auf, das in
Saudi-Arabien und anderen Ländern des Nahen Ostens
eine Reihe hoch pathogener Infektionen der Atemwege
verursachte [29]. Trotz der Befürchtungen
beschleunigte sich der Ausbruch 2013 nicht und die
Fälle von MERS-CoV beliefen sich auf 855 Personen
und 333 Todesfälle, was zu einer Sterblichkeitsrate
von fast 40% führte [23]. MERS-CoV ist ein
β-Coronavirus, das in hohem Maße mit den
Fledermaus-Coronaviren HKU4 und HKU5 verwandt ist
[32], was auf seinen Ursprung in Fledermäusen
hinweist, obwohl Dromedarkamele als Zwischenwirt
angegeben wurden [33]. Darüber hinaus wurde gezeigt,
dass sich MERS-CoV in Kamelzelllinien repliziert,
was weiter darauf hinweist, dass Kamele natürliche
Wirte für das Virus sein können [34]. Zusätzlich
zeigte eine Fallstudie, dass das aus einer Person,
die mit einem infizierten Kamel in Kontakt gekommen
war, isolierte Virus mit dem aus dem Kamel
isolierten Virus identisch war [35]. Im Gegensatz zu
SARS-CoV wird der Dipeptidylpeptidase 4 (DPP4)
-Rezeptor verwendet, wodurch Fledermäuse, Menschen,
Kamele, Kaninchen und Pferde anfällig werden [36].
Unterschiede in der Struktur des Maus-DPP4-Rezeptors
machen Mauszellen jedoch resistent gegen MERS-CoV,
was keine Bewertung von Virostatika und Impfstoffen
in geeigneten Mausmodellen ermöglicht. Ein
Mausmodell für MERS-CoV wurde jedoch durch
Einführung des menschlichen DPP4-Gens in die Lunge
der Maus durch einen adenoviralen Vektor konstruiert
[37].
Das derzeitige 2019-nCoV, offiziell als schweres
akutes respiratorisches Virus-Coronavirus-2
(SARS-CoV-2) bezeichnet, wurde erstmals im Dezember
2019 in der chinesischen Stadt Wuhan nachgewiesen
[38, 39]. Es wurde angenommen, dass es von
Wildtieren auf dem Huanan-Markt in Wuhan stammt und
sich durch Übertragung von Mensch zu Mensch
verbreitet, was die Krankheit COVID-19 mit
unterschiedlichem Schweregrad verursacht, von
leichten grippeähnlichen Symptomen bis hin zu
Lungenentzündung und Tod [ 39]. Fledermäuse,
Schlangen und Pangoline sind potenzielle Träger von
SARS-CoV-2, basierend auf dem Sequenzvergleich mit
Viren, die aus SARS-CoV-2-infizierten Patienten
isoliert wurden. Die COVID-19-Pandemie hat sowohl
SARS als auch MERS bereits in ihrer Schwere überholt
und kürzlich zu extremen Quarantänemaßnahmen
geführt, darunter die Abschottung großer Städte, die
Schließung von Grenzen und die Beschränkung der
Menschen auf ihre Häuser. Trotz dieser Bemühungen
gab es am 28. April 3.041.912 Fälle von COVID-19 und
211.167 Todesfälle in 210 Ländern [40]. Ob der Trend
zu einer weiteren Ausweitung der Pandemie führen
wird und wann sie aussterben oder in einem
saisonalen Muster zurückkehren wird, ist derzeit
nicht zu bestimmen, und nur die Zeit wird das
Ergebnis bestimmen.
Derzeit sind keine antiviralen Medikamente oder
Impfstoffe zur Behandlung von COVID-19 verfügbar
[41]. Der dringende Behandlungsbedarf hat daher eine
Vielzahl von Ansätzen zur Entwicklung neuartiger
Arzneimittel- und Impfstoffstrategien gegen COVID-19
ausgelöst. Zu den therapeutischen Ansätzen gehörten
Angiotensinrezeptorblocker, monoklonale Antikörper
(mAbs), Gen-Silencing und sogar Pflanzen- und
Pilzextrakte auf der Grundlage der traditionellen
chinesischen Medizin. Darüber hinaus umfasst die
Impfstoffentwicklung verschiedene Alternativen,
einschließlich epitopbasierter Peptidimpfstoffe,
viraler vektorbasierter Impfstoffe und der
Unterstützung von Bioinformatik und Immunoinformatik
für das Design effizienterer Ziele. Bevor die
verschiedenen Ansätze im Detail beschrieben werden,
ist es jedoch angebracht, einen Überblick über die
genomische Zusammensetzung von SARS-CoV-2 und seinen
Lebenszyklus zu geben, um potenzielle Arzneimittel-
und Impfstoffziele zu identifizieren.
2. Coronavirus-Genom und Lebenszyklus
Coronaviren besitzen ein nicht segmentiertes
Einzelstrang-RNA-Genom (ssRNA) mit positivem Sinn
von ungefähr 30 kb [42, 43]. Es enthält eine
5'-Endkappenstruktur, Replikase-Gene (rep 1a und rep
1b), die für die nichtstrukturellen Proteine
(nsPs) kodieren, die zwei Drittel des Genoms
umfassen, und die strukturellen Proteingene S
(Spike), E (Hülle), M (Membran) und N (Nucleocapsid)
sowie verschiedene akzessorische Gene, die in die
Strukturgene am 3'-Ende eingestreut sind [2]
(Abbildung 1). Obwohl die akzessorischen Proteine
als nicht essentiell für die Replikation angesehen
wurden, spielen sie eine wichtige Rolle bei der
viralen Pathogenese [43].

Abbildung 1. Schematische Darstellung des
Genoms des schweren akuten respiratorischen Syndroms
Coronavirus (SARS-CoV) -2. ORF1a und ORF1b codieren
die nichtstrukturellen Proteine. Die
Strukturproteine werden von Spike- (S), Hüll- (E),
Membran- (M) und Nucleocapsid- (N) Genen codiert.
Die Virionstruktur wurde durch
Kryo-Elektronentomographie und Mikroskopie
untersucht und zeigt die markanten keulenförmigen
Spike-Projektionen auf kugelförmige Partikel mit
einem Durchmesser von ca. 125 nm [44, 45]. Das
N-Protein ist helixsymmetrisch, wobei sowohl N- als
auch C-terminale Domänen für eine optimale
Einkapselung des RNA-Genoms benötigt werden [46].
Die Transkriptionsregulationssequenzen (TRS) [47]
und das genomische Verpackungssignal [48] wurden
identifiziert - von denen gezeigt wurde, dass
letzteres spezifisch an die C-terminale
RNA-Bindungsdomäne bindet, was die Verpackung des
Genoms in Viren erleichtert Partikel [49]. Das
M-Protein, das drei Transmembrandomänen enthält,
kann als Dimer vorliegen, bindet an das N-Protein
[50] und soll dem Virion seine Form geben [51]. Die
Topologie des kleinen 8–12 kD E-Proteins ist nicht
vollständig aufgelöst, es wird jedoch angenommen,
dass es eine Transmembranstruktur mit einer
N-terminalen Ektodomäne und einer C-terminalen
Endodomäne mit Ionenkanalaktivität aufweist [52]. Es
erleichtert den Aufbau und die Freisetzung von
Virionen und wurde mit der Pathogenese in Verbindung
gebracht [53]. Die aus dem S-Protein bestehenden
Spikes verleihen dem Virion das Aussehen einer
Sonnenkorona und vermitteln die Bindung an den
Wirtsrezeptor [54]. Das S-Protein wird für die
meisten Coronaviren durch eine Furin-ähnliche
Protease der Wirtszelle in S1- und S2-Polypeptide
gespalten [55, 56]. Bei SARS-CoV und SARS-verwandtem
CoV fehlt jedoch die Furinspaltungsstelle, während
SARS-CoV-2 diese beherbergt [57]. Ein fünftes
Strukturprotein, die Hämagglutininesterase (HE), ist
im Genom einiger Coronaviren wie MHV vorhanden [58].
Es wird angenommen, dass die Funktion des HE die
Verbesserung des S-Protein-vermittelten
Zelleintritts und der Ausbreitung über die
Schleimhaut [59] sowie die Erhöhung der
Neurovirulenz von MHV [60] ist.
Der Coronavirus-Lebenszyklus ist durch verschiedene
Schritte gekennzeichnet, einschließlich Anheften und
Eintreten, Expression von Replikase-Proteinen,
Replikation und Transkription sowie Assemblierung
und Freisetzung reifer Viruspartikel (Abbildung 2).
Die anfängliche Bindung erfolgt zwischen der
Rezeptorbindungsdomäne (RBD) der S1-Region des
S-Proteins und seinem Rezeptor. MHV verwendet eine
RBD am N-Terminus [61], während SARS-CoV eine RBD am
C-Terminus der S1-Region aufweist [62]. Die
S-Protein-Rezeptor-Wechselwirkung steuert den
Virustropismus, der auch das Targeting verschiedener
Rezeptoren durch verschiedene Coronaviren
widerspiegelt. Beispielsweise zielt MHV auf das
karzinoembryonale Antigen-verwandte Adhäsionsmolekül
1 (CEACAM1) [63], die MERS-CoV-Dipeptidylpeptidase 4
(DPP4) [64] sowie SARS-CoV und HCoVNL63 ACE2 [65]
ab.
Kürzlich wurde gezeigt, dass SARS-CoV-2 auch ACE2
verwendet [21]. Nach der anfänglichen Anlagerung
erfolgt eine säureabhängige proteolytische Spaltung
des S-Proteins durch Cathepsin oder eine andere
Protease, gefolgt von einer Fusion in angesäuerten
Endosomen oder an der Plasmamembran, was zur
Freisetzung des viralen Genoms in das Zytoplasma
führt [66, 67]. .
Abbildung 2. Lebenszyklus von SARS-CoV-2. Die
Anlagerung erfolgt bei ACE2, gefolgt von der
Freisetzung von viraler RNA in das Zytoplasma. Der
Replikase (REP) -Komplex ist für die RNA-Replikation
verantwortlich. RNA und das translatierte
Nucleocapsid (N) -Protein bilden Nucleocapside,
während die Spike- (S), Hüll- (E) und
Membranproteine (M) das
ER-Golgi-Zwischenkompartiment (ERGIC) und Golgi
durchlaufen, bevor die Viruspartikel zusammengesetzt
werden auf der Plasmamembran, von wo aus reife
Virionen freigesetzt werden.
Die Translation der Coronavirus-Replikase-Gene aus
zwei großen offenen Leserahmen (ORFs), rep1a und
rep1b, ermöglicht die Expression von zwei
Polyproteinen, pp1a und pp1ab, unter Verwendung
einer rutschigen Sequenz (5'-UUUAAAC-3 ') und eines
RNA-Pseudoknoten [ 68,69]. Die komplizierte
Zusammensetzung der pp1a- und pp1ab-Polyproteine
und der Aufbau von nsPs werden von Fehr und
Perlman ausführlich beschrieben [2]. Nach der
Translation und Assemblierung des
Replikase-Komplexes findet eine virale RNA-Synthese
sowohl aus genomischen als auch aus subgenomischen
RNAs statt. Letzteres dient als mRNA für die
Struktur- und Nebengene [2]. In der nicht
translatierten 5'-Region gibt es cis-wirkende
Sequenzen wie Stamm-Schleifen-Strukturen, die für
die Replikation viraler RNA wichtig sind [70, 71,
72, 73]. Ein interessantes Merkmal der
Coronavirus-Replikation betrifft die Fusion der
Leader- und Body-TRS-Segmente während der Produktion
subgenomischer RNAs. Obwohl ursprünglich angenommen
wurde, dass es während der Positivstrangsynthese
auftritt, legt ein neueres Modell nahe, dass die
RNA-abhängige RNA-Polymerase (RdRp) an einer der
TRS-Sequenzen des Körpers während der
diskontinuierlichen Verlängerung der
Negativstrang-RNA pausiert, gefolgt von einer
fortgesetzten Verlängerung zum nächsten TRS oder
Umschalten auf Amplifikation der Leadersequenz am
5'-Ende [74, 75].
Im Rahmen der Assemblierung werden die viralen
Strukturproteine S, E und M translatiert und über
Sekretionswege in das endoplasmatische Retikulum
(ER) und das ER-Golgi-Zwischenkompartiment (ERGIC)
transportiert [76, 77]. Am ERGIC sind virale Genome,
die von N-Protein umgeben sind, an der Bildung
reifer Virionen beteiligt [78]. Zusammengesetzte
Virionen werden dann in Vesikeln zur Zelloberfläche
transportiert und durch Exozytose freigesetzt [79].
Eine interessante Beobachtung betrifft Coronaviren,
die ohne das S-Protein zusammengesetzt wurden, was
zur Bildung von riesigen mehrkernigen Zellen führt,
die das Virus innerhalb eines infizierten Organismus
verbreiten, ohne von virusspezifischen Antikörpern
nachgewiesen oder neutralisiert zu werden [2].
3. Therapeutische und
prophylaktische Optionen
Seit dem SARS-CoV-Ausbruch in den Jahren 2002–2003
wurden zahlreiche Ansätze zur Entwicklung von
Therapeutika und Impfstoffen gegen Coronaviren
verwendet (Tabelle 2). Die jüngste COVID-19-Pandemie
hat die Bemühungen um Heilung und Schutz weiter
beschleunigt. Typischerweise werden drei allgemeine
Methoden zur antiviralen Behandlung von Coronaviren
angewendet [80]: (i) Anwendung bestehender
antiviraler Breitbandmedikamente; (ii) Screening von
chemischen Bibliotheken, die viele vorhandene
Verbindungen oder Datenbanken enthalten; (iii)
Neuentwicklung neuer spezifischer Arzneimittel auf
der Grundlage des Genoms und des biophysikalischen
Verständnisses einzelner Coronaviren. Weitere
Optionen sind monoklonale Antikörper und virale
Rezeptorblocker [81]. Es wurden auch Produkte
vorgeschlagen, die als traditionelle chinesische
Arzneimittel entwickelt wurden [80]. Im Einklang mit
gentherapeutischen Anwendungen wurde das Design
verschiedener Gen-Silencing-Ansätze auf der
Grundlage von RNA-Interferenz (RNAi) initiiert [82].
Im Zusammenhang mit der Impfstoffentwicklung wurde
eine Vielzahl von Immunisierungsstrategien
eingeleitet [83].
3.1. Therapeutika
Unter den Therapeutika hat die Verwendung der
Proteaseinhibitoren Lopinavir und Ritonavir als
Kombinationstherapie mit anderen antiretroviralen
Mitteln zur Behandlung von HIV-1-Infektionen zu
einer dauerhaften virologischen Unterdrückung und
verbesserten immunologischen Ergebnissen geführt
[84]. Die Kombinationstherapie mit Lopinavir und
Ritonavir wurde bei Krallenaffen mit einer
MERS-ähnlichen Erkrankung untersucht [85]. Im
Vergleich zu Mycophenolatmofetil (MFF) zeigte die
Behandlung verbesserte klinische, radiologische und
pathologische Ergebnisse und niedrigere mittlere
Viruslasten in nekropsierten Lungen- und
extrapulmonalen Geweben, während mit MFF behandelte
Tiere schwere oder tödliche Erkrankungen
entwickelten. Basierend auf diesen Ergebnissen wurde
ein Studienprotokoll für eine klinische Studie für
hospitalisierte erwachsene MERS-Patienten erstellt
[86]. Ziel ist es, laborbestätigte MERS-Patienten in
einer rekursiven, zweistufigen, multizentrischen,
placebokontrollierten, doppelblinden,
randomisierten, kontrollierten Studie zu
untersuchen. Im Zusammenhang mit der jüngsten
COVID-19-Pandemie wurde eine klinische Studie an 99
Patienten, die Lopinavir und Ritonavir erhielten,
mit 100 Patienten verglichen, die einer
Standardversorgung unterzogen wurden. Es wurde
jedoch kein Unterschied in der klinischen
Verbesserung, Mortalität oder nachweisbaren viralen
RNA erhalten [87]. In einer modifizierten
Intention-to-Treat-Analyse war die mediane Zeit bis
zur klinischen Besserung für die Behandlung mit
Lopinavir / Ritonavir einen Tag kürzer, und obwohl
gastrointestinale unerwünschte Ereignisse häufiger
auftraten, waren schwerwiegende unerwünschte
Ereignisse weniger häufig. Insgesamt ergab die
Studie, dass die Behandlung im Vergleich zur
Standardversorgung keinen Nutzen bot. Ein anderer
Ansatz umfasst die Anwendung von Nukleosidanaloga
als antivirale Mittel [88]. Beispielsweise wurden
mehrere Klassen von Nukleosidanaloga gegen SARS-CoV
in Vero-Zellen verifiziert [89]. In der Studie
zeigte das D-Isomer des Thyminanalogons eine starke
Anti-SARS-CoV-Aktivität und zeigte bei der höchsten
getesteten Dosierung von 100 uM keine Toxizität.
Obwohl das L-Cytosin-Analogon eine gute Aktivität
zeigte, zeigte es eine starke Toxizität für Zellen.
Ebenso lieferte das 3'-Azido-2 ', 3'-ungesättigte
Thyminanalogon eine starke Anti-SARS-CoV-Aktivität,
aber auch eine signifikante Toxizität. Zusätzlich
zeigte Dioxalan-Thymin eine mäßige antivirale
Aktivität ohne signifikante Zytotoxizität. Obwohl
C-Nucleosid, 4-Amino-7- (β-L-ribofuranosylpyrrolo
[3,2-D] pyrimidinhydrochlorid, die
SARS-CoV-Replikation inhibierte, war es zytotoxisch.
Insgesamt zeigten mehrere Klassen von
Nucleosidanaloga gegen SARS-CoV ein moderates
Ergebnis In vitro konnte keine eindeutige
Struktur-Aktivitäts-Beziehung hergestellt werden. In
einer anderen Studie wurde eine Reihe von doppelt
flexiblen Nukleosidanaloga auf der Basis des
Acrylzuckergerüsts von Aciclovir entworfen [90].
Eine Verbindung zeigte eine selektive antivirale
Aktivität gegen HCoV-NL63 und MERS-CoV. Im Gegensatz
dazu wurde keine Aktivität gegen SARS-CoV
nachgewiesen. In einer Studie zu FIP wurde das
Nucleosidanalogon GS-441524, ein Vorläufer des
pharmakologisch aktiven
Nucleosidtriphosphatmoleküls, als alternatives
Substrat und RNA-Kettenabschluss verwendet von
viralem RdRp war nicht toxisch und hemmte wirksam
die FIPV-Replikation in Katzen-CRFK-Zellen [91].
Darüber hinaus zeigten alle zehn mit GS-441524
behandelten Katzen in zwei Wochen eine rasche
Umkehrung der Krankheitssymptome Bei einem anderen
Ansatz wurde das Guanosinanalogon Ribavirin zusammen
mit Interferon-α2a bei erwachsenen Patienten mit
laborbestätigter MERS-CoV-Infektion und
Lungenentzündung verabreicht, die eine
Beatmungsunterstützung benötigten [92].
Die Studie zeigte, dass Patienten, die mit Ribavirin
und Interferon-α2a behandelt wurden, nach 14 Tagen
eine signifikant verbesserte Überlebensrate zeigten,
jedoch nicht nach 28 Tagen Behandlung im Vergleich
zur Kontrollgruppe der Patienten. Das
Adenosin-Nucleosid-Analogon Remdesivir hat sich als
wirksam zur Hemmung von RNA-Viren wie Filoviren,
Pneumoviren und Paramyxoviren durch Targeting von
RdRp erwiesen [93].
Darüber hinaus wurde die antivirale Aktivität von
Remdesivir auch für endemische und zoonotische
Coronaviren des Menschen wie HCoV-OC43 und HCoV-229E
nachgewiesen [94]. In einer anderen Studie wurde
gezeigt, dass Remdesivir RdRp von MERS-CoV wirksam
hemmt [95]. Kürzlich wurde gezeigt, dass Remdesivir
SARS-CoV-2 in Vero-Zellen und in menschlichen
Huh-7-Leberkrebszellen, von denen bekannt ist, dass
sie für SARS-CoV-2 anfällig sind, wirksam hemmt
[96]. Der erste Fall einer mitfühlenden Behandlung
von COVID-19 mit intravenöser Verabreichung von
Remdesivir fand in den USA statt, was zu einer
Verbesserung des Zustands des Patienten mit einem
Rückgang der Viruslast führte [97].
Neuraminidase-Hemmer wie Zanamivir, Laninamivir,
Oseltamivir und Peramivir haben Wirksamkeit gegen
die meisten Influenzastämme gezeigt [98]. In diesem
Zusammenhang wurden die Neuraminidase-Inhibitoren
Oseltamivir (Tamiflu) und Zanamivir (Relenza) in
einem cytopathischen Endpunkt-Assay in Vero-Zellen
auf die Hemmung der zytopathischen Wirkungen von
SARS-CoV getestet [99]. Keiner der
Neuraminidase-Inhibitoren zeigte jedoch eine Wirkung
auf SARS-CoV. Arbidol, ein antivirales
Breitbandmittel gegen verschiedene DNA- und
RNA-Viren, wurde in Russland und China zur
Vorbeugung und Behandlung von Influenza zugelassen
[100]. In Vero-Zellen wurde eine antivirale Wirkung
gegen das Zika-Virus (ZIKV), das West-Nil-Virus
(WNV) und das durch Zecken übertragene
Enzephalitis-Virus (TBEV) nachgewiesen [100].
Darüber hinaus wurde gezeigt, dass Arbidol sechs
verschiedene Isolate von ZIKV hemmen und vor den
zytopathischen Wirkungen von ZIKV schützen kann
[101].
Im Zusammenhang mit SARS-CoV wurden Patienten mit im
Labor bestätigtem COVID-19 mit oralem Arbidol in
Kombination mit einer Monotherapie mit Lopinavir /
Ritonavir oder Lopinavir / Ritonavir behandelt
[102]. Nach sieben Tagen konnte SARS-CoV in
nasopharyngealen Proben bei 12 von 16 Patienten, die
der Kombinationstherapie unterzogen wurden, nicht
nachgewiesen werden, verglichen mit 6 von 17 in der
Monotherapiegruppe. Die Zahlen am Tag 14 waren 15
von 16 für die Kombinationstherapie und 9 von 17 für
die Monotherapie am Tag 14. Darüber hinaus zeigten
CT-Scans eine Verbesserung bei 11 von 16 Patienten,
die die Kombinationstherapie erhielten, und 5 von
17, die die Monotherapie erhielten.
Tabelle 2. Therapeutische Interventionen gegen
Coronaviren.
Im Zusammenhang mit SARS-CoV wurden Patienten mit im
Labor bestätigtem COVID-19 mit oralem Arbidol in
Kombination mit einer Monotherapie mit Lopinavir /
Ritonavir oder Lopinavir / Ritonavir behandelt
[102]. Nach sieben Tagen konnte SARS-CoV in
nasopharyngealen Proben bei 12 von 16 Patienten, die
der Kombinationstherapie unterzogen wurden, nicht
nachgewiesen werden, verglichen mit 6 von 17 in der
Monotherapiegruppe. Die Zahlen am Tag 14 waren 15
von 16 für die Kombinationstherapie und 9 von 17 für
die Monotherapie am Tag 14. Darüber hinaus zeigten
CT-Scans eine Verbesserung bei 11 von 16 Patienten,
die die Kombinationstherapie erhielten, und 5 von
17, die die Monotherapie erhielten.
Tabelle 2. Therapeutische Interventionen
gegen Coronaviren.
Ein heißes Thema war heute die potenzielle
therapeutische Wirkung des weit verbreiteten
Arzneimittels gegen Malaria und
Autoimmunerkrankungen Chloroquin auf Coronaviren
[96]. Chloroquin blockiert die Virusinfektion und
stört die Glykosylierung der zellulären Rezeptoren
von SARS-CoV [110]. Die Anwendung von Chloroquin bei
COVID-19-Patienten wurde durch die jahrzehntelange
häufige Anwendung durch Reisende in geografischen
Regionen mit Malaria-Endemie und die kontinuierliche
Anwendung durch Einheimische gerechtfertigt [111].
Da jedoch Hinweise auf schwerwiegende Nebenwirkungen
des Einsatzes von Chloroquin berichtet wurden,
sollte Hydroxychloroquin in Betracht gezogen werden,
das eine ähnliche antivirale Wirksamkeit wie
Chloroquin besitzt, jedoch ein sichereres klinisches
Profil aufweist [112]. In einer begrenzten
klinischen Studie wurden 20 französische
COVID-19-Patienten mit Hydroxychloroquin behandelt,
was zu einer signifikanten Verringerung der
Viruslast im Vergleich zu Kontrollpatienten führte
[103]. Die Zugabe von Azithromycin verbesserte die
Effizienz der Viruselimination weiter signifikant.
Trotz der geringen Probengröße zeigte die Studie,
dass die Behandlung mit Hydroxychloroquin mit einer
signifikanten Verringerung der Viruslast und dem
Verschwinden von COVID-19 verbunden war. Das
Studiendesign war jedoch schlecht und die Ergebnisse
unzuverlässig, da sechs Patienten abbrachen und die
Beurteilung der Wirksamkeit auf der Viruslast
beruhte, die kein klinischer Endpunkt war. Daher
sind bei einer größeren Anzahl von Patienten
zusätzliche Studien erforderlich, um die Sicherheit
und Wirksamkeit von Hydroxychloroquin gründlich zu
validieren.
Die traditionelle chinesische Medizin hat auch
versucht, im Kampf gegen Coronaviren Unterstützung
zu leisten. In diesem Zusammenhang wurden
Fallstudien zur Behandlung mit der Shuanghuanglian
Oral Liquid (SHL) einer Familie mit COVID-19
berichtet [104]. Fall 1, eine 51-jährige Frau mit
hohem Fieber, bei der COVID-19 diagnostiziert wurde,
wurde fünf Tage später in eine Isolationsstation
eingewiesen, wo sie zweimal täglich mit der oralen
SHL begann. Am folgenden Tag wurde die
SHL-Verabreichung auf drei Dosen pro Tag erhöht,
ohne dass andere Medikamente eingenommen wurden. Das
Fieber nahm in zwei Tagen ab und sie erholte sich
allmählich. Fall 2, die 27-jährige Tochter von Fall
1, hatte hohes Fieber, Erbrechen und Durchfall. Zwei
Tage, nachdem sie in einer Isolationsstation
eingesperrt war, begann sie dreimal täglich mit der
Einnahme von SHL. Einige Tage später wurde eine
Abnahme der Körpertemperatur und ein erholter
Appetit festgestellt. Fall 3, der Ehemann von Fall
1, hatte leichten Durchfall, Erbrechen und Fieber
und wurde mit COVID-19 diagnostiziert. Er begann
dreimal täglich mit der Einnahme von SHL sowie
Moxifloxacin und Arbidol, was dazu führte, dass alle
Symptome mit Ausnahme der leichten Übelkeit
innerhalb von vier Tagen verschwanden. In einer
anderen Studie wurden vier Patienten mit leichter
oder schwerer 2019-nCoV-Pneumonie mit Lopinavir /
Ritonavir, Arbidol und traditioneller chinesischer
Medizin in Form von Shufeng Jiedu-Kapseln behandelt
[105]. Drei Patienten zeigten eine signifikante
Verbesserung in Bezug auf die
Lungenentzündungssymptome und testeten
2019-nCoV-negativ, und ein Patient mit schwerer
Lungenentzündung zeigte ebenfalls eine Verbesserung.
Ein weiterer möglicher Ansatz für die
COVID-19-Behandlung betrifft AHCC, einen
standardisierten Pilzextrakt auf α-Glucan-Basis aus
Lenintula edodes, der bei Menschen, die mit WNV,
Influenzavirus, Vogelgrippevirus, Hepatitis C-Virus,
Papillomavirus und Herpes infiziert sind, eine
Immunstimulation gezeigt hat Virus, Hepatitis
B-Virus und HIV [113]. Obwohl die Wirksamkeit von
AHCC für SARS-CoV-2 noch nicht verifiziert wurde,
könnte dies ein attraktiver alternativer Ansatz
sein. Ebenso haben sich Lianhuaqingwen-Kapseln als
wirksam zur Vorbeugung und Behandlung von
Atemwegsinfektionen durch Influenza A erwiesen
[114], wurden jedoch noch nicht für SARS-CoV-2
validiert.
Angesichts therapeutischer Strategien zur schnellen
Bekämpfung von SARS-CoV-2 zielt ein Ansatz darauf
ab, den Viruseintritt unter Verwendung einer
löslichen Version von ACE2 zu blockieren, die an
eine Immunglobulin-Fc-Domäne fusioniert ist [81].
Dieser Ansatz wird weitreichende neutralisierende
Antikörper hervorrufen und das Immunsystem
stimulieren. Die rekombinante Proteinexpression kann
zur schnellen Herstellung eines Arzneimittels zur
mitfühlenden Verwendung verwendet werden, während
formelle klinische Studien durchgeführt und
Impfstoffe entwickelt werden. Ein weiteres mit ACE2
verwandtes Ziel ist die Transmembranprotease Serin 2
(TMPRSS2), die am S-Protein-Priming beteiligt ist
und für die TMPRSS2-Inhibitoren möglicherweise für
die klinische Verwendung zur Blockierung des
Coronavirus-Eintritts zugelassen sind [106]. Es
wurde auch gezeigt, dass Seren von rekonvaleszenten
SARS-Patienten in der Lage sind, den SARS-CoV-2
S-gesteuerten Eintritt kreuzneutral zu machen [106].
Im Zusammenhang mit ACE2 ist die Bindungsaffinität
von SARS-CoV und SARS-CoV-2 zwar ähnlich, das
Vorhandensein der Furin-Spaltstelle im SARS-CoV-2
S-Protein unterscheidet sich jedoch durch das Design
spezifischer Inhibitoren [ 57]. Darüber hinaus
zeigte der Befund, dass polyklonale SARS-CoV
S-Maus-Antikörper den Eintritt von SARS-CoV-2
S-vermittelten Zellen wirksam verhindern können,
dass kreuzneutralisierende Antikörper gegen
konservierte S-Epitope ein praktikabler
Therapieansatz sein könnten. Im Zusammenhang mit
therapeutischen monoklonalen Antikörpern gegen
SARS-CoV-2 zeigte der erste SARS-CoV-spezifische
humane monoklonale Antikörper (mAb) CR3022 eine
starke Bindung an die
SARS-CoV-2-Rezeptorbindungsdomäne [107]. Das
CR3022-Epitop überlappt jedoch nicht mit der
ACE2-Bindungsstelle, was darauf hinweist, dass
CR3022 das Potenzial als Therapeutikum als solches
oder in Kombination mit anderen neutralisierenden
Antikörpern haben könnte. Interessanterweise konnten
die potenten SARS-CoV-spezifischen neutralisierenden
m396- und CR3014-Antikörper, die auf die
ACE2-Bindungsstelle abzielen, das SARS-CoV-2
S-Protein nicht binden, was auf Unterschiede in den
Rezeptorbindungsdomänen von SARS-CoV und SARS-CoV
hinweist -2. Es ist daher notwendig, mAbs mit
spezifischer Bindungsaffinität zur
SARS-CoV-2-Rezeptorbindungsdomäne zu entwickeln. Ein
weiterer vorläufiger Ansatz umfasst die Bewertung
bestehender Angiotensinrezeptor 1 (ATR1) -Blocker
wie Losartan, die üblicherweise zur Behandlung von
Bluthochdruck eingesetzt werden [108], um die
Aggressivität und Mortalität aufgrund von
SARS-CoV-2-Infektionen zu verringern [115]. Der
Ansatz basiert auf der Feststellung, dass ACE2
höchstwahrscheinlich die Bindungsstelle sowohl für
SARS-CoV als auch für SARS-CoV-2 darstellt und somit
ein vernünftiges Ziel für therapeutische
Interventionen bei Coronavirus-Infektionen
darstellt. Das Targeting von ATR1 könnte auch das
Problem mit neu auftretenden Coronavirus-Mutationen
lösen. Kürzlich wurde der erste Bericht über den
humanen 47D11-mAb veröffentlicht, der auf ein
konserviertes Epitop in der
Spike-Rezeptor-Bindungsdomäne abzielt [109]. Es
neutralisiert SARS-CoV und SARS-CoV-2 unabhängig von
der Hemmung der Rezeptorbindung und wird für die
Entwicklung von Antigen-Nachweis-Tests und
serologischen Tests nützlich sein. Darüber hinaus
erhöht es das Potenzial zur Vorbeugung und
Behandlung von COVID-19 und möglichen künftig
auftretenden Coronaviren.
3.2. Gen-Stummschaltung
Gen-Silencing auf Basis von RNA-Interferenz hat sich
als wichtiges Instrument in der Grundlagenforschung,
aber auch für therapeutische Anwendungen erwiesen
[116, 117]. Der Mechanismus von RNAi bezieht sich
auf doppelsträngige 19–23 Basenpaar-RNAs (dsRNAs),
die den Abbau von Ziel-RNA sequenzspezifisch
vermitteln [118]. Coronaviren wurden mehreren
Gen-Silencing-Studien unterzogen (Tabelle 3).
Beispielsweise wurde gezeigt, dass kurze
interferierende RNAs (siRNAs) die
SARS-CoV-Replikation in Vero E6-Zellen effizient
hemmen [119]. Darüber hinaus wurden 48
siRNA-Sequenzen im gesamten SARS-CoV-Genom
entworfen, die auf mehrere Schlüsselproteine
abzielen [120]. Chemisch synthetisierte siRNAs
wurden vor oder nach der SARS-CoV-Infektion in
fetale Rhesusnieren-FRhK4-Zellen transfiziert, und
die inhibitorischen Wirkungen wurden durch die
Abnahme der intrazellulären Kopienzahl des
Virusgenoms und der Virustiter verifiziert. Vier
siRNAs zeigten eine starke Hemmung der
SARS-CoV-Infektion und -Replikation. Prophylaktische
Wirkungen mit bis zu 90% Hemmung dauerten mindestens
72 Stunden. Die Kombination von siRNA-Duplexen aus
verschiedenen Regionen des viralen Genoms ergab eine
Hemmung von bis zu 80%. Darüber hinaus wurde
gezeigt, dass siRNA-Duplexe SARS-ähnliche Symptome
in vivo bei Rhesusaffen signifikant unterdrücken
[121]. In einem anderen Ansatz brachte die
Expression von U6-Promotor-gesteuerter siRNA, die
homolog zu ACE2-mRNA ist, die ACE2-Expression in
Vero-Zellen zum Schweigen [122]. Es wurde ferner
gezeigt, dass die SARS-CoV-Infektion in
ACE2-stillgelegten Zelllinien reduziert wurde, was
einen attraktiven Ansatz für siRNA-basierte
prophylaktische oder therapeutische Strategien
darstellt. Darüber hinaus wurden siRNA-Duplexe
eingesetzt, um die Expression des Aktin-bindenden
Proteins Ezrin, das während des Eintrittsstadiums
der Infektion mit dem SARS-CoV-Spike-Protein
interagiert, abzubauen [123].
Tabelle 3. Gen-Silencing gegen Coronaviren.
Im Zusammenhang mit MERS-CoV spielt die
ORF1ab-Region, die die Replikase-Polyproteine
codiert, eine entscheidende Rolle bei der
Virusinfektion und stellt daher ein geeignetes Ziel
für die Krankheitskontrolle dar. Vier miRNA- und
fünf siRNA-Moleküle wurden rational mithilfe von
Berechnungsmethoden zur Stummschaltung von neun
verschiedenen MERS-CoV-Stämmen entwickelt, um die
Behandlung von MERS-CoV auf genomischer Ebene zu
untersuchen [124]. Bei Versuchen, die Abgabe von
MERS-CoV-siRNAs zu optimieren, wurden Transfektion,
Elektroporation und virale Gentransfers angewendet.
Kürzlich wurde eine fortschrittliche Nanotechnologie
auf der Basis von Lipiden, Polymeren und
anorganischen Verbindungen formuliert [128]. Die
Gen-Stummschaltung von FIPV durch siRNAs wurde durch
Mutationen behindert, die resistente Viren erzeugen.
Um das Problem anzugehen, verhinderte die
Kombinationstherapie mit drei siRNAs das Entweichen
von Viren über fünf Passagen [125]. Darüber hinaus
lieferten Dicer-Substrat-siRNAs eine äquivalente
oder bessere Wirksamkeit als kanonische siRNAs für
FIPV. Im Zusammenhang mit Schweine-Deltacoronavirus
(PDCoV) wurden zwei kurze Haarnadel-RNA (shRNA)
exprimierende Plasmide, die auf die M- (pGenesil-M)
und N- (pGenesil-N) Gene von PDCoV abzielen, in
Schweinehodenzellen (ST) bewertet [126]. .
Herausforderungen mit dem PDCoV HB-BD-Stamm
ermöglichten einen hochspezifischen und effizienten
Schutz von ST-Zellen. Die Behandlung mit pGenesil-M
und pGenesil-N führte zu einer 13,2- bzw.
32,4-fachen Titerreduktion und einer Abnahme der
viralen RNA um 45,8% bzw. 56,1%. In einer anderen
Studie hemmten shRNAs, die auf das M-Gen des
Schweine-Epidemie-Durchfall-Virus (PEDV) und das
Schweine-Akon-Durchfall-Syndrom-Coronavirus
(SADS-CoV) und das N-Gen von PDCoV abzielen, die
Expression jeder viralen RNA über 98% [127]. Darüber
hinaus schränkten shRNAs die Expression von M- und
N-Proteinen signifikant ein und beeinträchtigten die
PEDV-, SADS-CoV- und PDCoV-Replikation.
3.3. Impfstoffentwicklung
Der klassische Ansatz für die Impfstoffentwicklung
gegen Viruserkrankungen umfasste die Immunisierung
mit abgeschwächten oder inaktivierten Lebendviren
[129]. Die Verfügbarkeit von Gentechnik und
effizienten Technologien zur Herstellung
rekombinanter Proteine hat die Anwendung auf die
Verwendung rekombinant exprimierter Antigene und
Immunogene zur Immunisierung verlagert [130]. Im
Zusammenhang mit Coronaviren begann die
Impfstoffentwicklung nach dem Ausbruch von SARS und
MERS ernsthaft und bot alternative Ansätze für die
Anwendung von Impfstoffen gegen Untereinheiten,
Impfstoffen für vollständig inaktivierte Viren,
Vektoren und abgeschwächte Lebendviren [83] (Tabelle
4). Es wurden auch Anstrengungen zur Bekämpfung
anderer Coronaviren untersucht. Beispielsweise wurde
ein modifizierter Lebendimpfstoff gegen BCoV durch
fortschreitende Abschwächung des respiratorischen
BCoV-Stammes 438/06-TN entwickelt [131]. Der
Impfstoff erwies sich als sicher und die
intramuskuläre Injektion bei Kälbern löste 30 Tage
nach der Impfung hohe Antikörpertiter gegen BCoV
aus. Das intranasale Medikament Bovilis® wurde gegen
durch BCoV verursachte Darmerkrankungen bei jungen
Kälbern zugelassen [132]. Das Fehlen relevanter
Impfstoffe gegen das Equine Coronavirus (ECoV)
katalysierte die Studie zu Antikörperreaktionen auf
ECoV bei Pferden nach Impfung mit dem BCoV-Impfstoff
[133]. Die Antikörpertiter gegen ECoV erhöhten sich
14 Tage nach der Inokulation bei allen sechs
geimpften Pferden, obwohl die Titer gegen ECoV
niedriger waren als gegen BCoV, und es blieb unklar,
ob die ausgelösten Antikörper Schutz gegen ECoV
boten.
Im Zusammenhang mit der Impfstoffentwicklung spielen
verschiedene Computer- und Informatikinstrumente
eine wesentliche Rolle. Beispielsweise wurde die
Immun-Epitop-Datenbank (IEDB) verwendet, um
geeignete MERS-CoV-Epitop-Impfstoffe gegen die
bekanntesten Allele der Weltbevölkerung auf der
Basis der S- und E-Proteine vorherzusagen [134].
Die Studie zeigte, dass hochkonservierte Sequenzen
in den S- und E-Proteinen als immunogen schützend
angesehen werden können und sowohl neutralisierende
Antikörper als auch T-Zell-Reaktionen hervorrufen
können, wenn sie mit B-Zellen,
T-Helferzell-Lymphozyten (HTLs) und zytotoxischen
T-Lymphozyten (CTLs) reagieren. In einem anderen
Ansatz wurde das SARS-CoV-2 S-Protein
charakterisiert, um immunogene Epitope für die
Impfstoffentwicklung zu erhalten [135]. Es wurden 13
Haupthistokompatibilitätskomplex (MHC) -I- und drei
MHC-II-Epitope mit antigenen Eigenschaften
identifiziert. Die Epitope wurden durch spezifische
Linker verknüpft und an den Toll-like-Rezeptor-5
(TLR5) angedockt, und die Immunoinformatik-Analyse
wurde für eine schnelle immunogene Profilierung
verwendet, um die Impfstoffentwicklung zu
beschleunigen. In einem anderen immunoinformatischen
und rechnerischen Ansatz wurden konservierte B- und
T-Zell-Epitope für das MERS-CoV S-Protein
identifiziert [136]. Die Antigenität der Epitope und
Wechselwirkungen mit dem B7-Allel des menschlichen
Leukozytenantigens (HLA) wurden geschätzt. Der
höchste Antigenitätswert wurde für das immens
immunogene B-Zell-Epitop QLQMGFGITVQYGT erhalten.
Die T-Zell-Epitoppeptide YKLQPLTFL (MHC-I) und
YCILEPRSG (MHC-II) waren ebenfalls stark antigen.
Diese identifizierten mutmaßlichen antigenen Epitope
könnten sich für die Entwicklung neuer Impfstoffe
als wirksam erweisen. Unter Verwendung von
In-Silico-Ansätzen wurden zwei
Multi-Epitop-Impfstoffe gegen MERS-CoV durch
Screening von CTL- und HTL-Epitopen aus 13
verschiedenen MERS-CoV-Proteinen entwickelt [137].
Beide Multi-Epitop-Impfstoffe trugen auch
potenzielle lineare B-Zell-Epitopregionen,
diskontinuierliche B-Zell-Epitope und
Interferon-γ-induzierende Epitope. Darüber hinaus
wurden menschliches β-Defensin-2 und β-Defensin-3
als Adjuvantien für verstärkte Immunantworten
verwendet. Die wirksamsten CTL- und HTL-Epitope und
Adjuvantien wurden durch kurze Peptidmolekularlinker
verbunden. Tertiärmodelle für beide
Multi-Epitop-Impfstoffe wurden auf ihre molekulare
Wechselwirkung mit TLR3 überprüft und cDNAs wurden
für die In-Silico-Analyse der Expression in
menschlichen Zelllinien erstellt, bevor sie in vivo
als potenzielle Impfstoffkandidaten getestet wurden.
Computergestützte Ansätze und Immunoinformatik haben
die oben beschriebene Impfstoffentwicklung stark
unterstützt. Darüber hinaus haben
Struktur-Funktions-Studien ebenfalls zu diesem Feld
beigetragen. In diesem Zusammenhang wurde im
Vergleich zu Antikörpern, die auf die
Rezeptorbindungsdomäne des MERS-CoV S-Proteins
abzielen, Antikörpern, die auf Epitope der
nichtrezeptorbindenden Domäne abzielen, wie dem
neutralisierenden Antikörper G2, der auf das
MERS-CoV abzielt, weniger Aufmerksamkeit geschenkt
N-terminale Domäne von S1 [138]. Die strukturelle
und funktionelle Charakterisierung von G2 allein
oder komplexiert mit dem MERS-CoV-N-Terminus von S1
zeigte, dass G2 die Bindung von MERS-CoV S an den
DDP4-Rezeptor stark stört und eine wichtige Rolle
als Ziel für die Immuntherapie und die
Impfstoffentwicklung spielen könnte. In einem
anderen Ansatz wurde die Kryo-Elektronenmikroskopie
(Kryo-EM) für das humane HCoV-NL63 angewendet, wobei
eine Auflösung des für den Viruseintritt in
Wirtszellen essentiellen Spike-Glykoprotein-Trimers
von 3,4 Å bereitgestellt wurde und das Hauptziel für
die Neutralisierung von Antikörpern darstellt [139].
Die Struktur enthüllte wichtige Komponenten des
Fusionsprozesses, einschließlich der Auslöseschleife
und der C-terminalen Domänen, die an der Verankerung
des Trimers an der Virusmembran beteiligt sind. Die
Studie ergab auch, dass HCoV-NL63 molekulare Tricks
verwendet, die auf der Epitopmaskierung mit Glykanen
und der Aktivierung von Konformationsänderungen bei
Versuchen beruhen, sich der Erkennung durch das
Immunsystem des Wirts zu entziehen. In einer anderen
Studie wurde das SARS-CoV-2 S-Protein einer
Kryo-EM-Strukturbestimmung des Ektodomänen-Trimers
unterzogen, um potenzielle Ziele für Impfstoffe und
die Hemmung des Viruseintritts zu identifizieren, da
die Furin-Spaltstelle zwischen den S1- und
S1-Untereinheiten einzigartig ist SARS-CoV-2 und
nicht in SARS-CoV oder anderen verwandten
Coronaviren vorhanden [57]. In einer Studie zu
SARS-CoV zeigte das Screening von experimentell
bestimmten Epitopen aus B- und T-Zellen aus den S-
und N-Proteinen, die identisch mit SARS-CoV-2
abgebildet sind, keine Mutationen unter den 120
verfügbaren SARS-CoV-2-Sequenzen [ 140]. Das
Immun-Targeting dieser Epitope kann daher
möglicherweise Schutz gegen SARS-CoV-2 bieten. In
Bezug auf die T-Zell-Epitope wurde eine
Populationsabdeckungsanalyse der assoziierten
MHC-Allele durchgeführt, die die Identifizierung
einer Reihe von Epitopen ermöglichte, die eine
breite globale Abdeckung bieten könnten.
Tabelle 4. Impfansätze gegen Voronaviren.
Impfstoffe auf Nukleinsäurebasis sind zu attraktiven
Alternativen zu Impfstoffen geworden, die auf
abgeschwächten oder inaktivierten Lebendviren
basieren. In diesem Zusammenhang wurden vier
Plasmid-DNA-basierte Impfstoffkonstrukte intradermal
an C57BL / 6-Mäuse verabreicht [141]. Der
pLL70-Vektor enthielt das SARS-CPoV S-Gen und der
pcDNA-SS-Vektor enthielt das codonoptimierte
SARS-CoV S-Gen, das mit der Leadersequenz aus dem
menschlichen CD5-Gen fusioniert war. Der
pcDNA-St-Vektor trug den N-Teil des codonoptimierten
S-Gens mit der CD5-Leadersequenz und der
pcDNA-St-VP22C enthielt den N-Teil des
codonoptimierten S mit der an das C- fusionierten
CD5-Leadersequenz. Terminal des VP22-Proteins von
Rinderherpesvirus-1 (BHV-1), von dem bekannt ist,
dass es die Proteinabgabe erleichtert und verbessert
[156]. Immunisierungsstudien zeigten, dass pcDNA-SS
und pcDNA-St-VP22C bei Mäusen überlegene zelluläre
und humorale Immunantworten hervorriefen und daher
die immunogensten SARS-Impfstoffkandidaten
darstellen. Darüber hinaus wurde der
DNA-Impfstoffansatz auf das SARS-CoV S-Protein unter
Verwendung eines verbesserten Plasmid-DNA-Vektors
verifiziert, der Donor- und Akzeptor-Spleißstellen
und heterologe virale RNA-Exportelemente wie das
konstitutive Transportelement (CTE) und das
posttranskriptionelle regulatorische Element ( PRE)
aus dem Mason-Pfizer-Affenvirus bzw. dem
Woodchuck-Hepatitis-Virus [142]. Diese
Vektormodifikationen verbesserten die Immunogenität
und Immunisierung von Mäusen mit 2 ug nackter
DNA-induzierter neutralisierender Anti-S-Antikörper
signifikant und boten Schutz gegen Herausforderungen
mit SARS-CoV.
In einer anderen auf Plasmid-DNA basierenden Studie
wurden Immunantworten auf einen nackten
Plasmidvektor und Plasmid /
Polyethylenimin-Nanopartikel, die das SARS-CoV
S-Protein exprimieren, in BALB / c-Mäusen nach
intranasaler Verabreichung untersucht [143]. Die
Immunisierung mit Nanopartikeln löste in der
Lungenwäsche signifikant höhere S-spezifische
IgG1-Antikörper in Seren und mukosalen
sekretorischen IgA-Antikörpern aus als in Mäusen,
die nackte Plasmid-DNA erhielten. In einer anderen
Studie wurde die DNA-Plasmid-basierte
Prime-Immunisierung mit einer Boost-Impfung mit
einem Protein kombiniert [144]. Truthähne wurden mit
einer oder zwei Dosen von 750 & mgr; g eines
DNA-Plasmids immunisiert, das ein
Truthahn-Coronavirus (TCoV) S-Proteinfragment trug,
das neutralisierende Epitope (4F / 4R) enthielt,
gefolgt von einem Boost mit 200 & mgr; g 4F /
4R-Fragment. Die Tiere wurden mit infektiösem TCoV
belastet und die klinischen Symptome wurden durch
einen Immunfluoreszenz-Antikörper-Assay überwacht.
Immunisierte Puten zeigten im Vergleich zu
Kontrolltieren weniger klinische Symptome und eine
geringere Viruslast. Die Impfung induzierte auch
humorale Immunantworten und bot einen teilweisen
Schutz gegen Herausforderungen mit TCoV.
Zelluläre und virale vektorbasierte
Expressionssysteme spielen eine wichtige Rolle bei
der Impfstoffentwicklung. Die Größe des CoV-Genoms
hat die Konstruktion infektiöser Klone zur
Verwendung in Studien über grundlegende virale
Prozesse und die Entwicklung genetisch definierter
Impfstoffe erschwert [156]. In diesem Zusammenhang
können bakterielle künstliche Chromosomen (BACs) ein
robustes System zur Expression von viraler RNA im
Zellkern unter der Kontrolle eines Cytomegalovirus
(CMV) -Promotors bereitstellen, gefolgt von einer
RNA-Amplifikation durch die virale Replikase im
Cytoplasma [157]. In einem prokaryotischen Ansatz
wurde ein eukaryotisches rekombinantes Plasmid, das
das SAD-Epitop (A- und D-Antigenstellen des
S-Proteins) von TGEV exprimiert, in Lactobacillus
acidophilus aus Schweinen transformiert [145]. Die
orale Verabreichung von L. acidophilus in BALB /
c-Mäusen induzierte signifikant höhere Spiegel an
S-IgA-Antikörpern im Vergleich zu einem
kommerziellen inaktivierten TGEV-Impfstoff. Die
Spiegel an TGEV-spezifischen IgGs waren ähnlich,
aber höhere Spiegel an Interferon-γ wurden durch den
L. acidophilus-Impfstoff induziert. Insgesamt
induzierte der orale TGEV L. acidophilus-Impfstoff
ein hohes Maß an mukosalen und humoralen
Immunantworten. In einem anderen Ansatz wurde eine
stabile CHO-Zelllinie konstruiert, um einen
rekombinanten Proteinimpfstoff der Untereinheit der
Reste 377-588 der Rezeptorbindungsdomäne des
MERS-CoV S-Proteins zu exprimieren [146]. Es wurde
gezeigt, dass der Impfstoff signifikante
neutralisierende Antikörperreaktionen hervorruft und
Schutz gegen MERS-CoV-Herausforderungen bei
geimpften Tieren bietet. Zur stabilen Expression
wurde das IL-2-Signalpeptid vor die an das humane
IgG-Fc-Fragment fusionierte S-Proteindomäne
eingeführt und in eine anhaftende CHO-Zelllinie mit
Dihydrofolatreduktase-Mangel transfiziert. Das
adCHO-exprimierte Fusionsprotein wurde sekretiert
und zeigte Funktionalität und Bindungsspezifität,
und eine Suspensions-CHO-Zelllinie wurde entwickelt.
Entwickelte transgene Mäuse mit dem für MERS-CoV
anfälligen DPP4-Rezeptor wurden mit dem Impfstoff
und Adjuvans der S377-588-Fc-Untereinheit
immunisiert, was zur Produktion neutralisierender
Antikörper gegen MERS-CoV und zum Überleben für
mindestens 21 Tage nach Herausforderungen mit
lebendem MERS- führte. CoV.
Ein interessanter Ansatz war die Verwendung von
Pflanzenexpressionssystemen für die Entwicklung von
Coronavirus-Impfstoffen. Beispielsweise wurde der
N-terminale Teil (Aminosäuren 1-658) des SARS-CoV
S-Proteins für die Codonverwendung in Pflanzen
optimiert und als Fusionsprotein mit dem grün
fluoreszierenden Protein (GFP) in Tabakblättern
exprimiert [147]. Es wurde gezeigt, dass das
S1-GFP-Fusionsprotein im Zytoplasma exprimiert
wurde. Eine stabile Expression des 35S-Promotors des
Blumenkohlmosaikvirus führte zu einer hohen
Expression des Fusionsproteins in Tabak- und
Salatblättern. Darüber hinaus wurde die
S1-Produktion auch in Chloroplasten-transformierten
Pflanzen erreicht, was auf das Potenzial für die
Entwicklung sicherer Impfstoffe gegen SARS-CoV aus
oralen pflanzlichen Untereinheiten hinweist. In
einer anderen Studie wurde ein N-terminales Fragment
des SARS-CoV S-Proteins in hohen Mengen in Tomaten-
und nikotinarmen Tabakpflanzen exprimiert [148]. Das
pflanzliche Antigen löste bei Mäusen systemische und
mukosale Immunantworten aus. In Mäusen wurden nach
oraler Verabreichung von Tomatenfrüchten, die das
S1-Protein exprimieren, signifikant erhöhte Spiegel
an SARS-CoV-spezifischen IgA-Antikörpern
nachgewiesen. Darüber hinaus wurden
SARS-CoV-spezifische IgG-Antikörper im Serum von
Mäusen nachgewiesen, die mit aus Tabak stammendem
S1-Protein grundiert waren. Eine verbesserte
Expression des SARS-CoV N-Proteins wurde in der
Tabakpflanze Nicotiana benthamiana erreicht, indem
das posttranskriptionelle
Gen-Silencing-Suppressor-p19-Protein aus dem
buschigen Stunt-Virus von Tomaten eingeschlossen
wurde [149]. Die intraperitoneale Verabreichung von
Pflanzenextrakt in BALB / c-Mäusen löste
N-Protein-spezifische IgG-Antikörper aus, und
insgesamt konnte geschlossen werden, dass die
pflanzliche Expression des SARS-CoV-N-Proteins bei
Mäusen starke humorale und zelluläre Immunantworten
induzieren kann. Schließlich haben die jüngsten
Fortschritte bei pflanzlichen Expressionssystemen
die Produktion zahlreicher Antigene und monoklonaler
Antikörper in Pflanzen ermöglicht [158], was nun
auch die Anwendung pflanzlicher Expressionssysteme
für die schnelle Impfstoffentwicklung gegen
SARS-CoV-2 beschleunigt hat.
Virale Vektoren wurden häufig als Abgabevehikel zur
Immunisierung gegen Infektionserreger wie pathogene
Viren verwendet [159]. In vielen Fällen wurden
starke humorale und zelluläre Immunantworten sowie
ein Schutz vor Herausforderungen mit tödlichen Dosen
pathogener Viren beobachtet. Im Zusammenhang mit
Coronaviren wurde ein rekombinanter
Adenovirusvektor, der das hitzelabile Enterotoxin B
(LTB) und das kernneutralisierende Epitop (COE) von
PEDV exprimierte, intramuskulär oder oral in BALB /
c-Mäuse und Ferkel verabreicht [150]. Drei Impfungen
in zweiwöchigen Intervallen erzeugten robuste
humorale und zelluläre Immunantworten.
Zellvermittelte Immunantworten wurden bei Mäusen
beobachtet und neutralisierende Antikörper hemmten
sowohl den Impfstoffstamm als auch neu auftretende
PEDV-Isolate. Bei Ferkeln wurden starke
Immunantworten beobachtet, es sind jedoch weitere
Studien erforderlich, um den Schutz vor
Herausforderungen mit hochvirulenten PEDV-Stämmen zu
überprüfen. In einer anderen Studie wurde der
modifizierte Vaccinia-Virus-Ankara (MVA) -Stamm zur
Expression des rekombinanten MERS-CoV S-Proteins
verwendet [151]. Die Immunisierung von
Dromedarkamelen löste eine Schleimhautimmunität aus.
Immunisierte Kamele zeigten eine signifikant
verringerte Ausscheidung von infektiösen Virus- und
viralen RNA-Transkripten, und der Schutz gegen
MERS-CoV korrelierte mit dem Vorhandensein
neutralisierender Antikörper im Serum. In einer
anderen Anwendung von MVA wurde das MERS-CoV N zur
Induktion zellulärer Immunantworten verwendet [152].
Die Identifizierung von MHC-I- und
MHC-II-beschränkten T-Zellantworten wurde an
überlappenden Peptiden durchgeführt, die das gesamte
MERS-CoV N-Polypeptid in BALB / c-Mäusen
überspannten, die mit MVA-MERS-CoV N immunisiert
waren. Ein H2-d-beschränktes Decamerpeptid Es wurde
ein Epitop identifiziert, das eine CD8 +
T-Zell-Antigenität zeigt, das weiteren Schutzstudien
in Mausmodellen für MERS-CoV unterzogen wird. Im
Zusammenhang mit SARS-CoV wurden die N- und
S-Protein-Gene zwischen den Glykoprotein-G- und
Polymerase-L-Genen in einem stark abgeschwächten
sicheren Tollwutvirus (RV) -Vektor kloniert [153].
Eine einzelne Injektion mit dem RV-SARS-CoV S-Vektor
induzierte bei Mäusen starke neutralisierende
Antikörperreaktionen, was ihn zu einem
vielversprechenden Kandidaten für die Ausrottung von
SARS-CoV aus Tierreservoirs macht.
Schließlich haben sich selbstreplizierende RNA-Viren
aufgrund der hocheffizienten Replikation ihres
ssRNA-Genoms mit positiver Polarität direkt im
Zytoplasma als Impfstoffvektoren als wirksam
erwiesen [159]. Da Vektoren entwickelt wurden, die
die Immunisierung von Plasmid-DNA-Replikons, nackten
RNA-Replikons oder replikationsdefizienten Partikeln
ermöglichen, ist der Anwendungsbereich breit.
Darüber hinaus sind aufgrund der Selbstreplikation
die Mengen, die erforderlich sind, um ähnliche
Immunantworten wie bei herkömmlichen DNA-Plasmiden
oder synthetischen mRNA-Molekülen zu erhalten, 100-
bis 1000-fach niedriger [160]. Im Zusammenhang mit
Coronaviren wurden Replikonpartikel des
venezolanischen Pferdeenzephalitis-Virus (VEE) zur
Expression der Urbani SARS-CoV S- oder N-Proteine
verwendet [154]. Es wurde gezeigt, dass
VEE-Partikel, die SARS-CoV S, aber nicht N
exprimieren, sowohl bei jungen als auch bei
seneszenten Mäusen einen vollständigen kurz- und
langfristigen Schutz gegen Herausforderungen mit
homologen Stämmen bieten. Um die Herausforderungen
heterologer Stämme zu bewerten, wurde ein chimäres
Virus (icGDO3-S) konstruiert, das ein synthetisches
S-Gen des genetisch divergierenden menschlichen
GDO3-Stammes codiert. Das chimäre Virus war gegen
Neutralisation mit Antiseren, die gegen den
Urbani-Stamm gerichtet waren, hochresistent. Die
Immunisierung mit VEE-Partikeln, die SARS-CoV S
exprimieren, bot jedoch einen vollständigen
Kurzzeitschutz gegen Herausforderungen mit icGDO3-S
bei jungen Mäusen, jedoch nicht bei seneszenten
Mäusen. Das Versagen, seneszierende Mäuse zu
schützen, wurde in einer anderen Studie
angesprochen, in der Mäuse mit VEE-Partikeln geimpft
wurden, die SARS-CoV S-Antigen exprimierten, das
entweder mit abgeschwächten (3014) oder Wildtyp
(3000) -VEE-Glykoproteinen verpackt war [155]. Die
Ergebnisse zeigten, dass gealterte Tiere, die mit
einem Impfstoff auf VEE (3000) -Basis immunisiert
waren, gegen SARS-CoV geschützt waren, während
Mäuse, die mit einem Impfstoff auf VEE (3014) -Basis
immunisiert waren, dies nicht waren. Darüber hinaus
wurde der überlegene Schutz auch für
Herausforderungen mit Influenzaviren bestätigt.
In Bezug auf die Sicherheit der Impfstoffentwicklung
umfasst die antikörperabhängige Verstärkung (ADE)
einen wichtigen Mechanismus, der den Eintritt
viraler Zellen erleichtert, wobei Virionen mit
Antikörpern komplexiert werden, was zu einer
verstärkten viralen Replikation führt [161]. Im
Zusammenhang mit COVID-19 kann eine vorherige
Infektion mit anderen Coronaviren, von Erkältung bis
SARS-CoV, COVID-19-Patienten vorbereitet haben und
eine schwere Erkrankung verursachen, sobald sie mit
SARS-CoV-2 infiziert sind. Dies könnte die
Diskrepanz zwischen der Schwere der Erkrankung bei
COVID-19-Patienten in der chinesischen Provinz Hubei
und anderen Regionen der Welt erklären [162].
Kürzlich wurde ein neuer molekularer Mechanismus für
ADE entdeckt, der zeigt, dass ein neutralisierender
Antikörper, der an das Coronavirus-S-Protein bindet,
eine Konformationsänderung des S-Proteins auslösen
kann, die den Viruseintritt über
IgG-Fc-Zellrezeptoren vermittelt [163]. Ein weiteres
Problem im Zusammenhang mit der Impfstoffentwicklung
betrifft die zelluläre Immunität vom Typ II gegen
Lungenpathogenität. In diesem Zusammenhang deuten
akkumulierte Hinweise darauf hin, dass die Lunge ein
wichtiger Ort der Immunregulation ist [164]. Daher
kann eine stark regulierte Immunantwort in der Lunge
vor einer Pathogeninfektion schützen. Im Gegensatz
dazu können ineffiziente Immunantworten verschiedene
Lungenerkrankungen auslösen. Im Zusammenhang mit der
SARS- und COVID-19-Lungenpathologie erfordert die
Genesung rigorose angeborene und erworbene
Immunantworten und epitheliale Regeneration [165].
Die Verabreichung von epithelialen Wachstumsfaktoren
wie dem Keratinozyten-Wachstumsfaktor (KGF) könnte
jedoch die Produktion von ACE2-exprimierenden Zellen
stimulieren und die Viruslast erhöhen.
Ein immuntherapeutischer Ansatz von großem Interesse
betrifft die Verwendung von Plasma von
Rekonvaleszenz-COVID-19-Patienten.
Rekonvaleszenzplasma wurde zuvor erfolgreich als
Postexpositionsprophylaxe und / oder Behandlung von
SARS und MERS eingesetzt [166]. Im Rahmen von
COVID-19 wurde in einer Fallstudie ein Patient mit
schwerem COVID-19 mit Rekonvaleszenzplasma von sechs
Spendern behandelt [167]. Die
Anti-SARS-CoV-IgM-Antworten aus dem
Rekonvaleszenzplasma waren schwach, es wurden jedoch
hohe IgG-Titer erhalten. Die Behandlung ermöglichte
es dem Patienten, 11 Tage nach der Plasmatransfusion
aus der mechanischen Beatmung entlassen und dann auf
eine allgemeine Station gebracht zu werden. In einer
anderen Fallstudie wurden fünf kritisch kranke
COVID-19-Patienten mit akutem Atemnotsyndrom (ARDS)
mit Rekonvaleszenzplasma behandelt, das von
Patienten erhalten wurde, die sich von COVID-19
erholt hatten [168]. Die Patienten, die mechanisch
beatmet wurden und mit antiviralen Mitteln und
Methylprednisolon behandelt worden waren, zeigten
innerhalb von 3 bis 4 Tagen eine normalisierte
Körpertemperatur, und ihre Viruslast nahm ab und
wurde innerhalb von 12 Tagen negativ. Darüber hinaus
erhöhten sich der SARS-CoV-2-spezifische ELISA und
die neutralisierenden Antikörpertiter, ARDS löste
sich bei vier Patienten nach 12 Tagen auf und drei
Patienten benötigten nach zwei Wochen keine
mechanische Beatmung. Drei Patienten wurden aus dem
Krankenhaus entlassen und die beiden verbleibenden
Patienten befanden sich in einem stabilen Zustand.
Offensichtlich erlauben die begrenzte Anzahl von
Patienten und das Studiendesign keine Bewertung der
Wirksamkeit der Behandlung, und weitere größere
klinische Studien sind erforderlich.
4. Schlussfolgerungen und
zukünftige Aspekte
Zusammenfassend gibt es derzeit keine wirksamen
antiviralen Medikamente oder Impfstoffe für
COVID-19. Aufgrund des Ausmaßes der aktuellen
COVID-19-Pandemie scheint sich jedoch die ganze Welt
zusammengeschlossen zu haben, um den Ausbruch zu
besiegen. Es scheint einen enormen politischen und
wirtschaftlichen Willen zu geben, Forschungs- und
Entwicklungsbemühungen auf beispiellose Weise zu
unterstützen. Das positive Ergebnis ist, dass alle
Wege untersucht werden, einschließlich antiviraler
Medikamente in Form bestehender Medikamente gegen
andere Viruserkrankungen, die einer gründlichen, gut
geplanten klinischen Bewertung unterzogen werden
sollten. Darüber hinaus werden neue Medikamente
gegen SARS-CoV-Rezeptoren in Form kleiner Moleküle
und monoklonaler Antikörper sowie
Gen-Silencing-Ansätze zur Verhinderung der
SARS-CoV-Replikation untersucht.
Da die Arzneimittelentwicklung und insbesondere die
für andere Indikationen verwendeten antiviralen
Arzneimittel eine Renaissance der klinischen Studien
für COVID-19 erlebt haben, ist es angebracht, die
jüngsten klinischen Ergebnisse zusammenzufassen. In
diesem Zusammenhang wurde Remdesivir bei 53
COVID-19-Patienten im Krankenhaus angewendet, von
denen 22 in den USA, 22 in Europa oder Kanada und 9
in Japan waren [169]. 36 der 53 Patienten (68%)
zeigten eine klinische Besserung, 25 Patienten
wurden entlassen und 7 starben. Zusätzlich wurden
COVID-19-Patienten in China einer klinischen Studie
für Remdesivir unterzogen [170]. Von den 237 in die
Studie eingeschlossenen Patienten erhielten 158
Remdesivir und 79 waren in der Placebogruppe. Die
Studie zeigte, dass Remdesivir nicht mit statistisch
signifikanten klinischen Vorteilen assoziiert war,
obwohl eine numerische Verkürzung der Zeit bis zur
klinischen Verbesserung beobachtet wurde. Am 29.
April 2020 veröffentlichte Gilead, der Hersteller
von Remdesivir, eine Pressemitteilung zu vorläufigen
Daten zu ihren SIMPLE-Studien der Phase III bei
COVID-19-Patienten [171]. Die erste der beiden
randomisierten, offenen, multizentrischen
SIMPLE-Studien an 397 Patienten zeigte, dass die
Zeit der klinischen Verbesserung bei 50% der
Patienten 10 Tage in der Gruppe betrug, die 5 Tage
lang intravenös Remdesivir erhielt, und 11 Tage nach
10 Tagen Behandlung im Vergleich Für die
Kontrollgruppe auf 15 Tage und mehr als die Hälfte
der Patienten in beiden Gruppen wurden bis zum 14.
Tag aus dem Krankenhaus entlassen. Es ist geplant,
die Studie zu erweitern, indem weitere 5600
Patienten aufgenommen und Studien an 180 Standorten
in China, Frankreich, durchgeführt werden.
Deutschland, Hongkong, Italien, Japan, Korea, die
Niederlande Singapur, Spanien, Schweden, die
Schweiz, Taiwan, das Vereinigte Königreich und die
Vereinigten Staaten. In der zweiten SIMPLE-Studie
wird die Sicherheit und Wirksamkeit der 5- und
10-tägigen Dosierungsdauer von intravenösem
Remdesivir mit der Standardbehandlung von
COVID-19-Patienten verglichen. Die ersten Ergebnisse
werden bis Ende Mai 2020 erwartet.
In Bezug auf Hydroxychloroquin wurde in Frankreich
eine erste klinische Bewertung bei 20 Patienten
durchgeführt, wie bereits beschrieben [106]. Obwohl
die Studie eine gewisse therapeutische Wirkung von
Hydroxychloroquin anzeigte und mindestens 80 Studien
mit Chloroquin, Hydroxychloroquin oder deren
Kombination mit anderen Arzneimitteln weltweit
registriert wurden, sollte man vorsichtig sein, da
viele Vorschläge auf In-vitro-Studien, Tiermodellen
oder Erfahrungen aus basieren andere
Viruserkrankungen [172]. Beispielsweise zeigte eine
chinesische Studie an mehr als 100 Patienten eine
Überlegenheit von Chloroquinphosphat im Vergleich
zur Kontrollbehandlung bei der Hemmung der
Exazerbation von Lungenentzündungen [173]. Darüber
hinaus wurden in einer placebokontrollierten
randomisierten Studie mit zwei verschiedenen
Hydroxychloroquin-Dosen bei 62 Patienten mit
radiologischer Lungenentzündungskonformation, jedoch
ohne schwere Hypoxie, nur für die höhere Dosis
geringfügige Verbesserungen der Körpertemperatur und
des Hustens registriert [174]. Die Ergebnisse der
niedrigeren Dosis wurden jedoch nicht beschrieben,
die im veröffentlichten Protokoll angegebenen
Endpunkte unterschieden sich von den angegebenen und
die Studie wurde vorzeitig abgebrochen [175].
Befürworter von Hydroxychloroquin haben auf die
Geschichte der breiten und sicheren Anwendung des
Arzneimittels hingewiesen. Hydroxychloroquin kann
Patienten jedoch bei gleichzeitiger Anwendung von
Azithromycin [178] möglicherweise schwerwiegenden
kutanen Nebenwirkungen [176], Leberversagen [177]
und ventrikulären Arrhythmien aussetzen [178].
Im Zusammenhang mit Lopinavir / Ritonavir zeigte
eine randomisierte, kontrollierte, offene Studie an
199 COVID-19-Patienten keinen Unterschied in Bezug
auf die klinische Verbesserung im Vergleich zur
Kontrollgruppe mit Standardversorgung [87]. Die
nachweisbaren viralen RNA-Spiegel und
Mortalitätszahlen waren für beide Gruppen ähnlich.
Gastrointestinale unerwünschte Ereignisse traten
häufiger bei Patienten auf, die mit Lopinavir /
Ritonavir behandelt wurden, schwerwiegende
unerwünschte Ereignisse traten jedoch in der
Standardversorgungsgruppe häufiger auf. Die
Wirksamkeit von Lopinavir Plus Ritonavir und Arbidol
gegen Coronavirus-Infektion (ELACOI) in einer
randomisierten kontrollierten Einzelblindstudie in
China umfasste 44 Patienten mit leichtem oder
mäßigem klinischen Status, die Lopinavir / Ritonavir
oder Arbidol (Umifenovir) einnahmen [179]. Die
Studie zeigte keine Unterschiede in der Zeit bis zum
negativen pharyngealen SARS-CoV-2-PCR-Nachweis,
Pyrexie, Husten oder Lungen-CT-Befunden zwischen der
Behandlungs- und Kontrollgruppe. In der Lopinavir /
Ritonavir-Gruppe verschlechterten sich jedoch 38,1%
der Patienten zu einem schweren / kritischen Status,
verglichen mit 12,5% und 14,3% in der Arbidol- bzw.
Kontrollgruppe. In der Arbidol- und Kontrollgruppe
wurden keine unerwünschten Ereignisse registriert.
Im Gegensatz dazu wurden nach einer Behandlung mit
Lopinavir / Ritonavir gastrointestinale und gestörte
unerwünschte Ereignisse in der Leber beobachtet.
Obwohl Medikamente, die für andere
Infektionskrankheiten entwickelt wurden, alternative
Behandlungsstrategien für COVID-19 bieten könnten,
wenn sie in gut konzipierten und gut durchgeführten
klinischen Studien evaluiert werden, scheint die
Entwicklung prophylaktischer Medikamente und
Impfstoffe, die auf bestimmte Strukturen von
SARS-CoV-2 abzielen, insgesamt bevorzugt zu sein
Ansatz. Im Zusammenhang mit der Impfstoffentwicklung
sollte die Verwendung von gereinigten Epitoppeptiden
und -antigenen sowie von zellulären oder viralen
Abgabesystemen parallel untersucht werden, um
maximalen Erfolg in einem minimalen Zeitrahmen zu
erzielen. Es überrascht nicht, dass mehr als 60
Impfstoffprojekte auf präklinischer oder klinischer
Ebene durchgeführt werden [180]. Diese Initiativen
wenden alle möglichen Verabreichungswege an,
einschließlich mRNA, Plasmid-DNA, nicht
replizierende virale Vektoren, inaktivierte und
lebende abgeschwächte Virus- und
Proteinuntereinheiten.
Es wird auch notwendig sein, die Möglichkeit
vorauszusehen, SARS-CoV-2-Ausbrüche in Zukunft zu
verhindern, und wenn nicht, besser auf eine zweite
oder dritte Welle von Coronaviren oder anderen
Pandemien vorbereitet zu sein. Es ist wichtig, den
Ursprung von SARS-CoV-2 zu verstehen, anstatt
Fledermäusen die Schuld für die Ursache der Pandemie
zu geben [181]. Bei mehreren Gelegenheiten wurde
darauf hingewiesen, dass Fledermäuse die einzigen
fliegenden Säugetiere sind. Wie falsch ist das?
Tatsächlich sind Menschen die echten fliegenden
Säugetiere, die mit immer schnellerer
Geschwindigkeit und Frequenz um die Welt fliegen.
Erst im vergangenen Jahr stiegen schätzungsweise 4,5
Milliarden Passagiere in den Himmel. Darüber hinaus
legen neuere Erkenntnisse zur Verteidigung von
Fledermäusen nahe, dass Pangoline die
Hauptverdächtigen als Quelle von SARS-CoV-2 sind,
obwohl dies noch nicht bestätigt wurde [182]. In
jedem Fall führen alle Wege zurück zum Menschen, da
Pangoline wegen ihres Fleisches und ihrer Schuppen
gefragt sind. Anstelle des Schuldspiels sollte der
Schwerpunkt nun auf beschleunigten Bemühungen zur
Entwicklung neuer sicherer und wirksamer
prophylaktischer und therapeutischer Ansätze in Form
von Coronavirus-Arzneimitteln und -Impfstoffen
liegen.
Finanzierung
Diese Forschung erhielt keine externe
Finanzierung.
Interessenskonflikte
Der Autor erklärt keinen Interessenkonflikt.
Verweise
- De Groot, R. J.; Baker, S. C.;
Baric, R.; Enjuanes, L.; Gorbalenya, A.
E.; Holmes, K. V.; Perlman, S.; Poon, L.;
Rottier, P. J.; Talbot, P. J.; et al.
Familie Coronaviridae. Im neunten Bericht
des Internationalen Komitees für Taxonomie
von Viren; Internationales Komitee für
Taxonomie von Viren, Internationale Union
mikrobiologischer Gesellschaften.
Abteilung Virologie; King, A. M.,
Lefkowitz, E., Adams, M. J., Carstens, E.
B., Eds.; Elsevier: Oxford,
Großbritannien, 2011; S. 806–828. ISBN
978-0-12-384684-6. [Google Scholar]
- Fehr, A. R.; Perlman, S.
Coronaviruses: Ein Überblick über ihre
Replikation und Pathogenese. Methoden Mol.
Biol. 2015, 1282, 1–23. [Google Scholar]
[PubMed]
- Doyle, L. P.; Hutchings, L. M.
Eine übertragbare Gastroenteritis bei
Schweinen. Marmelade. Tierarzt. Med.
Assoc. 1946, 108, 257–259. [Google
Scholar] [PubMed]
- Lin, C. M.; Saif, L. J.;
Marthaler, D.; Wang, Q. Evolution,
Antigenität und Pathogenität des
epidemischen Schweine-Durchfallvirus.
Virus Res. 2016, 226, 20–39. [Google
Scholar] [CrossRef] [PubMed]
- Mora-Díaz, J. C.; Piñeyro, P.
E.; Houston, E.; Zimmerman, J.;
Giménez-Lirola, L.G.
Schweinehämagglutinierendes
Enzephalomyelitis-Virus: Ein Rückblick.
Vorderseite. Tierarzt. Sci. 2019, 6, 53.
[Google Scholar] [CrossRef]
- Amer, H.M. Rinderähnliche
Coronaviren bei Haus- und
Wildwiederkäuern. Anim. Gesundheit Res.
Rev. 2018, 19, 113–124. [Google Scholar]
[CrossRef]
- Perlman, S.; Netland, J.
Coronaviruses nach SARS: Update zu
Replikation und Pathogenese. Nat. Rev.
Microbiol. 2009, 7, 439–450. [Google
Scholar] [CrossRef]
- Funk, C. J.; Manzer, R.; Miura,
T. A.; Groshong, S. D.; Ito, Y.; Travanty,
E. A.; Leete, J.; Holmes, K. V.; Mason,
R.J. Atemwegs-Coronavirus-Infektion bei
Ratten: Replikation in Atemwegs- und
Alveolarepithelzellen und angeborene
Immunantwort. J. Gen. Virol. 2009, 90,
2956–2964. [Google Scholar] [CrossRef]
- Cavanagh, D. Coronavirus
Vogelinfektiöses Bronchitis-Virus.
Tierarzt. Res. 2007, 38, 281–297. [Google
Scholar] [CrossRef]
- Wolfe, L. G.; Griesemer, R.A.
Infektiöse Peritonitis bei Katzen. Pathol.
Tierarzt. 1966, 3, 255–270. [Google
Scholar] [CrossRef]
- Jaimes, J. A.; Whittaker, G.A.
Feline Coronavirus: Einblicke in die
virale Pathogenese basierend auf der
Struktur und Funktion des Spike-Proteins.
Virology 2018, 517, 108–121. [Google
Scholar] [CrossRef]
- Izumi, T. Sarkoidose in verschiedenen
Organen. Clin. Dermatol. 1986, 4, 150–153.
[Google Scholar] [CrossRef]
- Mihindukulasuriya, K. A.; Wu, G.; St
Leger, J.; Nordhausen, R. W.; Wang, D.
Identifizierung eines neuartigen Coronavirus
aus einem Beluga-Wal unter Verwendung eines
panviralen Microarrays. J. Virol. 2008, 82,
5084–5088. [Google Scholar] [CrossRef]
[PubMed]
- He, B.; Zhang, Y.; Xu, L.; Yang, W.; Yang,
F.; Feng, Y.; Xia, L.; Zhou, J.; Zhen, W.;
Feng, Y.; et al. Identifizierung
verschiedener Alphacoronaviren und
genomische Charakterisierung eines
neuartigen Coronavirus wie Fledermäuse in
China. J. Virol. 2014, 88, 7070–7082.
[Google Scholar] [CrossRef] [PubMed]
- Menachery, V. D.; Yont, B. L., Jr.;
Debbink, K.; Agnihothram, S.; Gralinski, L.
E.; Plante, J. A.; Graham, R. L.; Scobey,
T.; Ge, X. Y.; Donaldson, E. F.; et al. Ein
SARS-ähnlicher Cluster von
Fledermaus-Coronaviren zeigt das Potenzial
für die Entstehung beim Menschen. Nat. Med.
2015, 21, 1508–1513. [Google Scholar]
[CrossRef]
- Wege, H.; Siddell, S. G.; ter Meulen, V.
Die Biologie und Pathogenese von
Coronaviren. Curr. Top Microbiol. Immunol.
1982, 99, 165–200. [Google Scholar]
- Lampert, P. W.; Sims, J. K.; Kniazeff,
A.J. Mechanismus der Demyelinisierung bei
JHM-Virus-Enzephalomyelitis. Acta
Neuropathol. 1973, 24, 76–85. [Google
Scholar] [CrossRef]
- Bradburne, A. F.; Bynoe, M. L.; Tyrell,
D.A.J. Auswirkungen eines „neuen“
menschlichen Atemwegsvirus bei Freiwilligen.
Br. Med. J. 1967, 3, 767–769. [Google
Scholar] [CrossRef]
- van der Hoek, L.; Sicher, K.; Ihorst, G.;
Stang, A.; Pyrc, K.; Jebbink, M. F.;
Petersen, G.; Forster, J.; Berkhout, B.;
Uberla, K. Croup ist mit dem neuartigen
Coronavirus NL63 assoziiert. PLoS Med. 2005,
2, e240. [Google Scholar] [CrossRef]
- Hamre, D.; Procknow, J.J. Ein neues Virus,
das aus den menschlichen Atemwegen isoliert
wurde. Proc. Soc. Exp. Biol. Med. 1966, 121,
190–193. [Google Scholar] [CrossRef]
- Anderson, R. M.; Fraser, C.; Ghani, A. C.;
Donelly, C. A.; Riley, S.; Ferguson, N. M.;
Leung, G. M.; Lam, T. H.; Hedley, A.J.
Epidemiologie, Übertragungsdynamik und
Kontrolle von SARS: Die Epidemie 2002–2003.
Philos. Trans. R Soc. London B Biol. Sci.
2004, 359, 1091–1105. [Google Scholar]
[CrossRef]
- Cherry, J. D. Die Chronologie der
SARS-Minipandemie 2002–2003. Pädiatrie. Bzw.
Rev. 2004, 5, 262–269. [Google Scholar]
[CrossRef] [PubMed]
- Aleanizi, F. S.; Mohmed, N.; Alqahtani, F.
Y.; El Hadi Mohamed, R.A. Ausbruch des
Coronavirus des respiratorischen Syndroms im
Nahen Osten in Saudi-Arabien: Eine
retrospektive Studie. BMC-Infektion. Dis.
2017, 17, 23. [Google Scholar] [CrossRef]
[PubMed]
- Guan, Y.; Zheng, B. J.; He, Y.Q.; Liu, X.
L.; Zhuang, Z. X.; Cheung, C. L.; Luo, S.
W.; Li, P. H.; Zhang, L. J.; Guan, Y. J.; et
al. Isolierung und Charakterisierung von
Viren im Zusammenhang mit dem
SARS-Coronavirus aus Tieren in Südchina.
Science 2003, 302, 276–278. [Google Scholar]
[CrossRef] [PubMed]
- Lau, S. K.; Woo, P. C.; Li, K. S.; Huang,
Y.; Tsoi, H. W.; Wong, B. H.; Wong, S. S.;
Leung, S. Y.; Chan, K. H.; Yuen, K.Y.
Schweres akutes respiratorisches Syndrom
Coronavirus-ähnliches Virus bei chinesischen
Hufeisenfledermäusen. Proc. Natl. Acad. Sci.
USA 2005, 102, 14040–14045. [Google Scholar]
[CrossRef]
- Li, W.; Shi, Z.; Yu, M.; Ren, W.; Smith,
C.; Epstein, J. H.; Wang, H.; Crameri, G.;
Hu, Z.; Zhang, H.; et al. Fledermäuse sind
natürliche Reservoire von SARS-ähnlichen
Coronaviren. Science 2005, 310, 676–679.
[Google Scholar] [CrossRef]
- Ge, X. Y.; Li, J. L.; Yang, X. L.; Chmura,
A. A.; Zhu, G.; Epstein, J. H.; Mazet, J.
K.; Hu, B.; Zhang, W.; Peng, C.; et al.
Isolierung und Charakterisierung eines
Fledermaus-SARS-ähnlichen Coronavirus, das
den ACE2-Rezeptor verwendet. Nature 2013,
503, 535–538. [Google Scholar] [CrossRef]
- Peiris, J. S.; Yuen, K. Y.; Osterhaus, A.
D.; Stohr, K. Das schwere akute
respiratorische Syndrom. N. Engl. J. Med.
2003, 349, 2431–2441. [Google Scholar]
[CrossRef]
- Zaki, A. M.; van Boheemen, S.; Bestebroer,
T. M.; Osterhaus, A. D.; Fouchier, R.A.
Isolierung eines neuartigen Coronavirus von
einem Mann mit Lungenentzündung in
Saudi-Arabien. N. Engl. J. Med. 2012, 367,
1814–1820. [Google Scholar] [CrossRef]
- van Boheemen, S.; de Graaf, M.; Lauber,
C.; Bestebroer, T. M.; Raj, V. S.; Zaki, A.
M.; Osterhaus, A. D.; Haagmans, B. L.;
Gorbalenya, A. E.; Snijder, E. J.; et al.
Genomische Charakterisierung eines neu
entdeckten Coronavirus im Zusammenhang mit
dem akuten Atemnotsyndrom beim Menschen.
mBio 2012, 3, e00473-12. [Google Scholar]
[CrossRef]
- Chibo, D.; Birch, C. Die Analyse der
Spike- und Nucleoprotein-Gene des humanen
Coronavirus 229E zeigt eine genetische Drift
zwischen chronologisch unterschiedlichen
Stämmen. J. Gen. Virol. 2006, 87, 1203–1208.
[Google Scholar] [CrossRef]
- Vijgen, L.; Keyaerts, E.; Lemey, P.; Moes,
E.; Li, S.; Vandamme, A. M.; Van Ranst, M.
Zirkulation genetisch unterschiedlicher
zeitgenössischer humaner
Coronavirus-OC43-Stämme. Virology 2005, 337,
85–92. [Google Scholar] [CrossRef] [PubMed]
- Meyer, B.; Muller, M. A.; Corman, V. M.;
Reusken, C. B.; Ritz, D.; Godeke, G. J.;
Lattwein, E.; Kallies, S.; Siemens, A.; van
Beek, J.; et al. Antikörper gegen
MERS-Coronavirus bei Dromedarkamelen,
Vereinigte Arabische Emirate, 2003 und 2013.
Emerg. Infizieren. Dis. 2014, 20, 552–559.
[Google Scholar] [CrossRef] [PubMed]
- Eckerle, I.; Corman, V. M.; Muller, M. A.;
Lenk, M.; Ulrich, R. G.; Drosten, C.
Replikationskapazität des MERS-Coronavirus
in Tierzelllinien. Emerg. Infizieren. Dis.
2014, 20, 276–279. [Google Scholar]
[CrossRef] [PubMed]
- Azhar, E. I.; El-Kafrawy, S. A.; Farraj,
S. A.; Hassan, A. M.; Al-Saeed, M. S.;
Hashem, A. M.; Madani, T.A. Hinweise auf die
Übertragung des MERS-Coronavirus von Kamel
zu Mensch. N. Engl. J. Med. 2014, 370,
2499–2505. [Google Scholar] [CrossRef]
- Raj, V. S.; Mou, H.; Smits, S. L.;
Dekkers, D. H.; Muller, M. A.; Dijkman, R.;
Muth, D.; Demmers, J. A.; Zaki, A.;
Fouchier, R. A.; et al. Dipeptidylpeptidase
4 ist ein funktioneller Rezeptor für das
aufkommende humane Coronavirus-EMC. Nature
2013, 495, 251–254. [Google Scholar]
[CrossRef]
- Zhao, J.; Li, K.; Wohlford-Lenane, C.;
Agnihothram, S. S.; Fett, C.; Zhao, J.;
Gale, M. J., Jr.; Baric, R. S.; Enjuanes,
L.; Gallagher, T.; et al. Schnelle
Generierung eines Mausmodells für das
Atmungssyndrom im Nahen Osten. Proc. Natl.
Acad. Sci. USA 2014, 111, 4970–4975. [Google
Scholar] [CrossRef]
- Kaplan, E.H. Enthält 2019-nCoV (Wuhan)
Coronavirus. Gesundheitsmanagement Sci.
2020. [Google Scholar] [CrossRef]
- Yang, Y.; Peng, F.; Wang, R.; Guan, K.;
Jiang, T.; Xu, G.; Sun, J.; Chang, C. Das
tödliche Coronavirus; Die SARS-Epidemie 2003
und die neuartige Coronavirus-Epidemie 2020
in China. J. Autoimmun. 2020. [Google
Scholar] [CrossRef]
- Globale Gesundheitspolitik. Online
verfügbar:
https://www.google.com/covid19-map (Zugriff
am 28. April 2020).
- Han, Q.; Lin, Q.; Jin, S.; Sie, L.
Coronavirus 2019-nCoV: Eine kurze
Perspektive von der Front. J. Infect. 2020.
[Google Scholar] [CrossRef]
- Kahn, J.; McIntosh, K. Geschichte und
jüngste Fortschritte bei der Entdeckung von
Coronaviren. Ped. Infizieren. Dis. J. 2005,
24, s223 - s227. [Google Scholar] [CrossRef]
- Zhao, L.; Jha, B. K.; Wu, A.; Elliott, R.;
Ziebuhr, J.; Gorbalenya, A. E.; Silverman,
R. H.; Weiss, S.R. Der Antagonismus des
Interferon-induzierten OAS-RNase L-Weges
durch das murine Coronavirus ns2-Protein ist
für die Virusreplikation und die
Leberpathologie erforderlich. Zelle. Host
Microbe 2012, 11, 607–616. [Google Scholar]
[CrossRef] [PubMed]
- Barcena, M.; Oostergetel, G. T.;
Bartelink, W.; Faas, F. G.; Verkleij, A.;
Rottier, P. J.; Koster, A. J.; Bosch, B. J.
Kryo-Elektronentomographie des
Maus-Hepatitis-Virus: Einblicke in die
Struktur des Coronavirions. Proc. Natl.
Acad. Sci. USA 2009, 106, 582–587. [Google
Scholar] [CrossRef] [PubMed]
- Neuman, B. W.; Adair, B. D.; Yoshioka, C.;
Quispe, J. D.; Orca, G.; Kuhn, P.; Milligan,
R. A.; Yeager, M.; Buchmeier, M. J.
Supramolekulare Architektur des Coronavirus
des schweren akuten respiratorischen
Syndroms, nachgewiesen durch
Elektronenkryomikroskopie. J. Virol. 2006,
80, 7918–7928. [Google Scholar] [CrossRef]
[PubMed]
- de Groot, R. J.; Luytjes, W.; Horzinek, M.
C.; van der Zeijst, B. A.; Spaan, W. J.;
Lenstra, J.A. Hinweise auf eine
Coiled-Coil-Struktur in den Spike-Proteinen
von Coronaviren. J. Mol. Biol. 1987, 196,
963–966. [Google Scholar] [CrossRef]
- Stohlman, S. A.; Baric, R. S.; Nelson, G.
N.; Soe, L. H.; Welter, L. M.; Deans, R.J.
Spezifische Wechselwirkung zwischen
Coronavirus-Leader-RNA und
Nucleocapsid-Protein. J. Virol. 1988, 62,
4288–4295. [Google Scholar] [CrossRef]
[PubMed]
- Molenkamp, R.; Spaan, W. J.
Identifizierung einer spezifischen
Wechselwirkung zwischen dem
Nukleokapsidprotein des
Coronavirus-Maus-Hepatitis-Virus A59 und dem
Verpackungssignal. Virology 1997, 239,
78–86. [Google Scholar] [CrossRef]
- Kuo, L.; Masters, P.S. Funktionsanalyse
des genomischen RNA-Verpackungssignals des
murinen Coronavirus. J. Virol. 2013, 87,
5182–5192. [Google Scholar] [CrossRef]
- Neuman, B. W.; Kiss, G.; Kunding, A. H.;
Bhella, D.; Baksh, M. F.; Connelly, S.;
Droese, B.; Klaus, J. P.; Makino, S.;
Sawicki, S. G.; et al. Eine Strukturanalyse
des M-Proteins in der
Coronavirus-Assemblierung und -Morphologie.
J. Struct. Biol. 2011, 174, 11–22. [Google
Scholar] [CrossRef]
- Armstrong, J.; Niemann, H.; Smeekens, S.;
Rottier, P.; Warren, G. Sequenz und
Topologie eines Modell-intrazellulären
Membranproteins, E1-Glykoprotein, aus einem
Coronavirus. Nature 1984, 308, 751–752.
[Google Scholar] [CrossRef]
- Godet, M.; L'Haridon, R.; Vautherot, J.
F.; Laude, H. TGEV-Koronavirus ORF4 codiert
ein Membranprotein, das in Virionen
eingebaut ist. Virology 1992, 188, 666–675.
[Google Scholar] [CrossRef]
- Nieto-Torres, J. L.; Dediego, M. L.;
Verdia-Baguena, C.; Jimenez-Guardeno, J. M.;
Regla-Nava, J. A.; Fernandez-Delgado, R.;
Castano-Rodriguez, C.; Alcaraz, A.; Torres,
J.; Aguilella, V. M.; et al. Schwere
Coronavirus-Hüllprotein-Ionenkanalaktivität
mit akutem respiratorischem Syndrom fördert
die Fitness und Pathogenese des Virus. PLoS
Pathogens 2014, 10, 1004077. [Google
Scholar] [CrossRef] [PubMed]
- Collins, A. R.; Knobler, R. L.; Powell,
H.; Buchmeier, M. J. Monoklonale Antikörper
gegen das murine Hepatitis-Virus-4 (Stamm
JHM) definieren das virale Glykoprotein, das
für die Bindung und Zell-Zell-Fusion
verantwortlich ist. Virology 1982, 119,
358–371. [Google Scholar] [CrossRef]
- Abraham, S.; Kienzle, T. E.; Lapps, W.;
Brian, D.A. Abgeleitete Sequenz des
Rinder-Coronavirus-Spike-Proteins und
Identifizierung der internen proteolytischen
Spaltstelle. Virology 1990, 176, 296–301.
[Google Scholar] [CrossRef]
- Luytjes, W.; Sturman, L. S.; Bredenbeek,
P. J.; Charite, J.; van der Zeijst, B. A.;
Horzinek, M. C.; Spaan, W. J. Primärstruktur
des Glykoproteins E2 des Coronavirus MHV-A59
und Identifizierung der Trypsin-Spaltstelle.
Virology 1987, 161, 479–487. [Google
Scholar] [CrossRef]
- Walls, A. C.; Park, Y. J.; Tortorici, M.
A.; Wall, A.; McGuire, A. T.; Veesler, D.
Struktur, Funktion und Antigenität des
SARS-CoV-2-Spike-Glykoproteins. Zelle 2020.
[Google Scholar] [CrossRef] [PubMed]
- Klausegger, A.; Strobl, B.; Regl, G.;
Kaser, A.; Luytjes, W.; Vlasak, R.
Identifizierung einer
Coronavirus-Hämagglutininesterase mit einer
Substratspezifität, die sich von der des
Influenza-C-Virus und des Rinder-Coronavirus
unterscheidet. J. Virol. 1999, 73,
3737–3743. [Google Scholar] [CrossRef]
[PubMed]
- Cornelissen, L. A.; Wierda, C. M.; van der
Meer, F. J.; Herrewegh, A. A.; Horzinek, M.
C.; Egberink, H. F.; de Groot, R.J.
Hämagglutininesterase, ein neues
Strukturprotein des Torovirus. J. Virol.
1997, 71, 5277–5286. [Google Scholar]
[CrossRef]
- Kazi, L.; Lissenberg, A.; Watson, R.; de
Groot, R. J.; Weiss, S.R. Die Expression von
Hämagglutininesterase-Protein aus dem
rekombinanten Maus-Hepatitis-Virus verstärkt
die Neurovirulenz. J. Virol. 2005, 79,
15064–15073. [Google Scholar] [CrossRef]
- Kubo, H.; Yamada, Y. K.; Taguchi, F.
Lokalisierung neutralisierender Epitope und
der Rezeptorbindungsstelle innerhalb der
aminoterminalen 330 Aminosäuren des murinen
Coronavirus-Spike-Proteins. J. Virol. 1994,
68, 5403–5410. [Google Scholar] [CrossRef]
- Cheng, P. K.; Wong, D. A.; Tong, L. K.;
Ip, S. M.; Lo, A. C.; Lau, C. S.; Yeung, E.
Y.; Lim, W.W. Virusausscheidungsmuster des
Coronavirus bei Patienten mit wahrscheinlich
schwerem akutem respiratorischem Syndrom.
Lancet 2004, 363, 1699–1700. [Google
Scholar] [CrossRef]
- Nedellec, P.; Dveksler, G. S.; Daniels,
E.; Turbide, C.; Chow, B.; Basile, A. A.;
Holmes, K. V.; Beauchemin, N. Bgp2, ein
neues Mitglied der Genfamilie der
karzinoembryonalen Antigene, codiert einen
alternativen Rezeptor für
Maus-Hepatitis-Viren. J. Virol. 1994, 68,
4525–4537. [Google Scholar] [CrossRef]
[PubMed]
- van Doremalen, N.; Miazgowicz, K. L.;
Milne-Price, S.; Bushmaker, T.; Robertson,
S.; Scott, D.; Kinne, J.; McLellan, J. S.;
Zhu, J.; Munster, V.J.
Wirtsspeziesrestriktion des Coronavirus des
respiratorischen Syndroms im Nahen Osten
durch seine Rezeptor-Dipeptidylpeptidase 4.
J. Virol. 2014, 88, 9220–9232. [Google
Scholar] [CrossRef] [PubMed]
- Li, W.; Moore, M. J.; Vasilieva, N.; Sui,
J.; Wong, S. K.; Bern, M. A.; Somasundaran,
M.; Sullivan, J. L.; Luzuriaga, K.;
Greenough, T. C.; et al. Das
Angiotensin-Converting-Enzym 2 ist ein
funktioneller Rezeptor für das
SARS-Coronavirus. Nature 2003, 426, 450–454.
[Google Scholar] [CrossRef] [PubMed]
- Bosch, B. J.; van der Zee, R.; de Haan, C.
A.; Rottier, P. J. Das
Coronavirus-Spike-Protein ist ein
Klasse-I-Virus-Fusionsprotein: Strukturelle
und funktionelle Charakterisierung des
Fusionskernkomplexes. J. Virol. 2003, 77,
8801–8811. [Google Scholar] [CrossRef]
- Belouzard, S.; Chu, V. C.; Whittaker, G.R.
Aktivierung des
SARS-Coronavirus-Spike-Proteins durch
sequentielle proteolytische Spaltung an zwei
verschiedenen Stellen. Proc. Natl. Acad.
Sci. USA 2009, 106, 5871–5876. [Google
Scholar] [CrossRef]
- Baranov, P. V.; Henderson, C. M.;
Anderson, C. B.; Gesteland, R. F.; Atkins,
J. F.; Howard, M.T. Programmierte ribosomale
Frameshifting bei der Dekodierung des
SARS-CoV-Genoms. Virology 2005, 332,
498–510. [Google Scholar] [CrossRef]
- Brierley, I.; Digard, P.; Inglis, S. C.
Charakterisierung eines effizienten
ribosomalen Frameshifting-Signals des
Coronavirus: Voraussetzung für einen
RNA-Pseudoknoten. Cell 1989, 57, 537–547.
[Google Scholar] [CrossRef]
- Brown, C. G.; Nixon, K. S.; Senanayake, S.
D.; Brian, D.A. Eine RNA-Stammschleife
innerhalb der kodierenden Region des
Rinder-Coronavirus nsp1 ist ein
cis-wirkendes Element bei der fehlerhaften
störenden RNA-Replikation. J. Virol. 2007,
81, 7716–7724. [Google Scholar] [CrossRef]
- Guan, B. J.; Wu, H. Y.; Brian, D.A. Eine
optimale cis-Replikationsstammschleife IV in
der 5'-untranslatierten Region des
Maus-Coronavirus-Genoms erweitert 16
Nukleotide in den offenen Leserahmen 1. J.
Virol. 2011, 85, 5593–5605. [Google Scholar]
[CrossRef]
- Liu, P.; Kleine.; Keane, S. C.; Yang, D.;
Leibowitz, J. L.; Giedroc, D.P. Die
Stammschleife 2 des Hepatitis-Virus der Maus
nimmt eine Uynmg (U) a-ähnliche
Tetraloop-Struktur an, die gegenüber
Basensubstitutionen hochfunktionell tolerant
ist. J. Virol. 2009, 83, 12084–12093.
[Google Scholar] [CrossRef]
- Madhugiri, R.; Fricke, M.; Marz, M.;
Ziebuhr, J. Coronavirus cis-wirkende
RNA-Elemente. Adv. Virus Res. 2016, 96,
127–163. [Google Scholar] [PubMed]
- Raman, S.; Bouma, P.; Williams, G. D.;
Brian, D.A. Die Stammschleife III in der
5'-untranslatierten Region ist ein
cisaktives Element bei der defekten
störenden RNA-Replikation des
Rinder-Coronavirus. J. Virol. 2003, 77,
6720–6730. [Google Scholar] [CrossRef]
[PubMed]
- Sethna, P. B.; Hofmann, M. A.; Brian, D.A.
Minusstrangkopien von replizierenden
Coronavirus-mRNAs enthalten Antileader. J.
Virol. 1991, 65, 320–325. [Google Scholar]
[CrossRef] [PubMed]
- Sawicki, S. G.; Sawicki, D. L.; Siddell,
S. G. Eine zeitgenössische Ansicht der
Coronavirus-Transkription. J. Virol. 2007,
81, 20–29. [Google Scholar] [CrossRef]
[PubMed]
- Krijnse-Locker, J.; Ericsson, M.; Rottier,
P. J. M .; Griffiths, G. Charakterisierung
des Knospungskompartiments des
Maus-Hepatitis-Virus: Der Nachweis, dass der
Transport vom RER zum Golgi-Komplex nur
einen vesikulären Transportschritt
erfordert. J. Cell Biol. 1994, 124, 55–70.
[Google Scholar] [CrossRef] [PubMed]
- Tooze, J.; Tooze, S.; Warren, G.
Replikation des Coronavirus MHV-A59 in
Sackzellen: Bestimmung der ersten
Knospungsstelle von Nachkommenvirionen. EUR.
J. Cell Biol. 1984, 33, 281–293. [Google
Scholar]
- de Haan, C. A.; Rottier, P. J. Molekulare
Wechselwirkungen beim Aufbau von
Coronaviren. Adv. Virus Res. 2005, 64,
165–230. [Google Scholar]
- Lu, H. Optionen für die medikamentöse
Behandlung des neuen Coronavirus 2019
(2019.nCoV). Biosci. Trends. 2020. [Google
Scholar] [CrossRef]
- Kruse, R. L. Therapeutische Strategien in
einem Ausbruchszenario zur Behandlung des
neuartigen Coronavirus mit Ursprung in
Wuhan, China. F1000Res 2020, 9, 72. [Google
Scholar] [CrossRef]
- Wu, C. J.; Chan, Y.L. Antivirale
Anwendungen von RNAi für Coronaviren. Exp.
Meinung. Investig. Drugs 2006, 15, 89–97.
[Google Scholar] [CrossRef]
- Enjuanes, L.; Zuniga, S.;
Castano-Rodriguez, C.; Gutierrez-Alvarez,
J.; Canton, J.; Sola, I. Molekulare Basis
der Coronavirus-Virulenz und
Impfstoffentwicklung. Adv. Virus Res. 2016,
95, 245–286. [Google Scholar]
- Croxtall, J. D.; Perry, C.M. Lopinavir /
Ritonavir: Eine Überprüfung seiner
Verwendung bei der Behandlung von
HIV-1-Infektionen. Drugs 2010, 70,
1885–1915. [Google Scholar] [CrossRef]
[PubMed]
- Chan, J. F.; Yao, Y.; Yeung, M. L.; Deng,
W.; Bao, L.; Jia, L.; Li, F.; Xiao, C.; Gao,
H.; Jep.; et al. Die Behandlung mit
Lopinavir / Ritonavir oder Interferon-β1b
verbessert das Ergebnis einer
MERS-CoV-Infektion in einem
nichtmenschlichen Primatenmodell des
Murmeltiers. J. Infect. Dis. 2015, 212,
1904–1913. [Google Scholar] [CrossRef]
[PubMed]
- Arabi, Y. M.; Alothman, A.; Balkhy, H. H.;
Al-Dawood, A.; Al Johani, S.; Al Harbi, S.;
Kojan, S.; Al Jeraisy, M.; Deeb, A. M.;
Assiri, A.M. Behandlung des Atemwegssyndroms
im Nahen Osten mit einer Kombination aus
Lopinavir-Ritonavir- und
Interferon-β1b-Studie (MIRACLE):
Studienprotokoll für eine randomisierte
kontrollierte Studie. Versuche 2018, 19, 81.
[Google Scholar] [CrossRef]
- Cao, B.; Wang, Y.; Wen, D.; Liu, W.; Wang,
J.; Fan, G.; Ruang, L.; Song, B.; Cai, Y.;
Wei, M.; et al. Eine Studie mit
Lopinavir-Ritonavir bei Erwachsenen, die mit
schwerem Covid-19 ins Krankenhaus
eingeliefert wurden. N. Engl. J. Med. 2020.
[Google Scholar] [CrossRef]
- Prujisssers, A. J.; Denison, M. R.
Nucleosidanaloga zur Behandlung von
Coronavirus-Infektionen. Curr. Meinung.
Virol. 2019, 35, 57–62. [Google Scholar]
[CrossRef]
- Chu, C. K.; Gadthula, S.; Chen, X.; Choo,
H.; Olgen, S.; Barnard, D. L.; Sidwell, R.
W. Antivirale Aktivität von Nukleosidanaloga
gegen SARS-Coronavirus (SARS-CoV). Antivir.
Chem. Chemother. 2006, 17, 285–289. [Google
Scholar] [CrossRef]
- Peters, H. L.; Jochmans, D.; de Wilde, A.
H.; Posthuma, C. C.; Snijder, E. J.; Neyts,
J.; Seley-Radtke, K.L. Design, Synthese und
Bewertung einer Reihe von acyclischen
Fleximernukleosidanaloga mit
Anti-Coronavirus-Aktivität. Bioorg. Med.
Chem. Lette. 2015, 25, 2923–2926. [Google
Scholar] [CrossRef]
- Murphy, B. G.; Perron, M.; Murakami, E.;
Bauer, K.; Park, Y.; Eckstrand, C.;
Liepnieks, M.; Pedersen, N. C. Das
Nucleosidanalogon GS-441524 hemmt das Virus
der infektiösen Katzenperitonitis (FIP) in
Gewebekulturen und in experimentellen
Katzeninfektionsstudien stark. Tierarzt.
Microbiol. 2018, 219, 226–233. [Google
Scholar] [CrossRef]
- Omrani, A. S.; Saad, M. M.; Baig, K.;
Bahloul, A.; Abdul-Matin, M.; Alaidaroos, A.
Y.; Almakhlafi, G. A.; Albarrak, M. M.;
Memish, Z. A.; Albarrak, A.M. Ribavirin und
Interferon alpha-2a bei schwerer
Coronavirus-Infektion mit Atemwegssyndrom im
Nahen Osten: Eine retrospektive
Kohortenstudie. Lancet Infect. Dis. 2014,
14, 1090–1095. [Google Scholar] [CrossRef]
- Lo, M. K.; Jordan, R.; Arvey, A.;
Sudhamsu, J.; Shrivastava-Ranjan, P.;
Hotard, A. L.; Flint, M.; McMullan, L. K.;
Siegel, D.; Clarke, M. O.; et al. GS-5734
und sein Elternnukleosidanalogon hemmen
Filo-, Pneumo- und Paramyxoviren. Sci. Rep.
2017, 7, 43395. [Google Scholar] [CrossRef]
[PubMed]
- Brown, A. J.; Won, J. J.; Graham, R. L.;
Dinnon, K. H., 3 .; Sims, A. C.; Feng, J.
Y.; Cihlar, T.; Denison, M. R.; Baric, R.
S.; Sheahan, T.P. Antivirales
Breitband-Remdesivir hemmt endemische und
zoonotische Deltacoronaviren des Menschen
mit einer stark divergierenden
RNA-abhängigen RNA-Polymerase. Antivirale
Res. 2019, 169, 104541. [Google Scholar]
[CrossRef] [PubMed]
- Gordon, C. J.; Tchesnokov, E. P.; Feng, J.
Y.; Porter, D. P.; Gotte, M. Die antivirale
Verbindung Remdesivir hemmt wirksam die
RNA-abhängige RNA-Polymerase aus dem
Coronavirus des Atmungssyndroms im Nahen
Osten. J. Biol. Chem. 2020. [Google Scholar]
[CrossRef] [PubMed]
- Wang, M.; Cao, R.; Zhang, L.; Yang, X.;
Liu, J.; Xu, M.; Shi, Z.; Hu, Z.; Zhong, W.;
Xiao, G. Remdesivir und Chloroquin hemmen
das kürzlich aufgetauchte neuartige
Coronavirus (2019-nCoV) in vitro wirksam.
Cell Res. 2020, 30, 269–271. [Google
Scholar] [CrossRef] [PubMed]
- Ko, W.-C.; Rolain, J.-M.; Lee, N.-Y.;
Chen, P. L.; Huang, C.-T.; Lee, P.-I.;
Hsueh, P.-R. Argumente für Remdesivir zur
Behandlung von SARS-CoV-2-Infektionen. Int.
J. Antimicrob. Agenten 2020. [Google
Scholar] [CrossRef] [PubMed]
- Laborda, P.; Wang, S. Y.; Voglmeir, J.
Influenza-Neuraminidase-Inhibitoren:
Synthetische Ansätze, Derivate und
biologische Aktivität. Molecules 2016, 21,
1513. [Google Scholar] [CrossRef]
- Tan, E. L. C .; Eong Ooi, E.; Lin, C.-Y.;
Cheng Tan, H.; Ling, A. E.; Lim, B.;
Stanton, L.W. Hemmung der
SARS-Coronavirus-Infektion in vitro mit
klinisch zugelassenen Arzneimitteln. Emerg.
Inf. Dis. 2004, 10, 581–586. [Google
Scholar] [CrossRef]
- Haviemik, J.; Stefanik, M.; Foitikova, M.;
Kali, S.; Tordo, N.; Rudolf, I.; Hubalek,
Z.; Eyer, L.; Ruzek, D. Arbidol
(Umifenovir): Ein antivirales
Breitspektrum-Medikament, das medizinisch
wichtige von Arthropoden übertragene
Flaviviren hemmt. Viren 2018, 10, 184.
[Google Scholar] [CrossRef]
- Fink, S. L.; Voitech, L.; Waggoner, J.;
Slivinski, N. S. J .; Jackson, K. J.; Wang,
R.; Khadka, S.; Luthra, P.; Basler, C. F.;
Polyak, S.J. Das antivirale Medikament
Arbidol hemmt das Zika-Virus. Sci. Rep.
2018, 8, 8989. [Google Scholar] [CrossRef]
- Deng, L.; Li, C.; Zeng, Q.; Liu, X.; Li,
X.; Zhang, H.; Hong, Z.; Xia, J. Arbidol
kombiniert mit LPV / r gegenüber LPV / r
allein gegen die Corona-Virus-Krankheit
2019: Eine retrospektive Kohortenstudie. J.
Infect. 2020. [Google Scholar] [CrossRef]
- Gautret, P.; Lagier, J.-C.; Parola, P.;
Hoang, V. T.; Meddeb, L.; Mailhe, M.;
Doudier, B.; Courjon, J.; Giordanengo, V.;
Vieira, E. V.; et al. Hydroxychloroquin und
Azothromycin zur Behandlung von COVID-19:
Ergebnisse einer offenen, nicht
randomisierten klinischen Studie. Int. J.
Antimicrob. Agenten 2020. [Google Scholar]
[CrossRef] [PubMed]
- Ni, L.; Zhou, L.; Zhou, M.; Zhao, J.;
Wang, D.W. Kombination aus westlicher
Medizin und traditioneller chinesischer
Patentmedizin bei der Behandlung eines
Familienfalls von COVID-19 in Wuhan.
Vorderseite. Med. 2020. [Google Scholar]
[CrossRef] [PubMed]
- Wang, Z.; Chen, X.; Lu, Y.; Chen, F.;
Zhang, W. Klinische Merkmale und
therapeutisches Verfahren für vier Fälle mit
neuartiger Coronavirus-Pneumonie im Jahr
2019, die eine kombinierte Behandlung nach
chinesischer und westlicher Medizin
erhalten. Biosci. Trends 2020, 14, 64–68.
[Google Scholar] [CrossRef] [PubMed]
- Hoffmann, M.; Kleine-Weber, H.; Schroeder,
S.; Krüger, N.; Herrler, T.; Erichsen, S.;
Schiergens, T. S.; Herrler, G.; Wu, N.;
Nitsche, A.; et al. Der Eintritt von
SARS-CoV-2-Zellen hängt von ACE2 und TMPRSS2
ab und wird durch einen klinisch
nachgewiesenen Proteaseinhibitor blockiert.
Zelle 2020. [Google Scholar] [CrossRef]
[PubMed]
- Tian, X.; Li, C.; Huang, A.; Xia, S.;
Lu, S.; Shi, Z.; Lu, L.; Jiang, S.; Yang,
Z.; Wu, Y.; et al. Potente Bindung des
neuartigen Coronavirus-Spike-Proteins 2019
durch einen SARS-Coronavirus-spezifischen
humanen monoklonalen Antikörper. Emerg.
Mircobes. Infizieren. 2020, 9, 382–385.
[Google Scholar] [CrossRef] [PubMed]
- Gurwitz, D. Angiotensinrezeptorblocker als
vorläufige SARS-CoV-2-Therapeutika. Drug
Dev. Res. 2020. [Google Scholar] [CrossRef]
- Wang, C.; Li, W.; Drabek, D.; Okba, N. M.
A .; van Haperen, R.; Osterhaus, A. D.; van
Kupperveld, F.; Haagmans, B. L.; Grosveld,
F.; Bosch, B.-J. Ein menschlicher
monoklonaler Antikörper, der die
SARS-CoV-Infektion blockiert. bioRxiv 2020.
Preprint. [Google Scholar] [CrossRef]
- Vincent, M. J.; Bergeron, E.; Benjannet,
S.; Erickson, B. R.; Rollin, P. E.; Ksiazek,
T. G.; Seidah, G. S.; Nichol, S.T.
Chlkoroquin ist ein starker Inhibitor der
SARS-Coronavirus-Infektion und -Verbreitung.
Virol. J. 2005, 2, 69. [Google Scholar]
[CrossRef]
- Colson, P.; Rolain, J.-M.; Raoult, D.
Chloroquine für das neuartige Coronavirus
SARS-CoV-2 von 2019. Int. J. Antimicrob.
Agents 2020, 55, 105923. [Google Scholar]
[CrossRef]
- Zhou, D.; Dai, S. M.; Tong, Q. COVID-19:
Eine Empfehlung zur Untersuchung der Wirkung
von Hydrochloroquin bei der Verhinderung von
Infektionen und Progressionen. J.
Antimicrob. Chemother. 2020, 114. [Google
Scholar] [CrossRef]
- Di Pierro, F.; Bertuccioli, A.; Cavecchia,
I. Mögliche therapeutische Rolle einer hoch
standardisierten Wirkstoffmischung aus
kultivierten Lentinula edodes mycelia (AHCC)
bei Patienten, die mit dem neuartigen
Coronavirus 2019 infiziert sind. Minerva.
Gastoenterol. Dietol. 2020. [Google Scholar]
[CrossRef]
- Ding, Y.; Zeng, L.; Li, R.; Chen, Q.;
Zhou, B.; Chen, Q.; Cheng, P. L.; Yutao, W.;
Zheng, J.; Yang, Z. Die chinesische
verschreibungspflichtige
Lianhuaqingwen-Kapsel übt durch die Hemmung
der Virusvermehrung eine
Anti-Influenza-Aktivität aus und
beeinträchtigt die Immunfunktion. BMC
Complement Altern. Med. 2017, 17, 130.
[Google Scholar] [CrossRef] [PubMed]
- Moen, M. D.; Wagstaff, A.J. Losartan: Eine
Überprüfung seiner Verwendung bei der
Reduzierung des Schlaganfallrisikos bei
Patienten mit Bluthochdruck und
linksventrikulärer Hypertrophie. Drugs 2005,
65, 2657–2674. [Google Scholar] [CrossRef]
[PubMed]
- Lee, R. C.; Feinbaum, R. L.; Ambros, V.;
Das heterochrone Gen C.-elegans lin-4
codiert kleine RNAs mit Antisense, das zu
lin-14 komplementär ist. Cell 1993, 75,
843–854. [Google Scholar] [CrossRef]
- Lundstrom, K. Mikro-RNA in der Krankheits-
und Gentherapie. Curr. Drug Discov. Technol.
2011, 8, 76–86. [Google Scholar] [CrossRef]
- Shi, Y. Mammalian RNAi für die Massen.
Trends Genet. 2003, 19, 9–12. [Google
Scholar] [CrossRef]
- Zhang, Y.; Zündete.; Fu, L.; Yu, C.; Li,
Y.; Xu, X.; Wang, Y.; Ning, H.; Zhang, S.;
Chen, W.; et al. Stummschaltung der
SARS-CoV-Spike-Proteinexpression in
kultivierten Zellen durch RNA-Interferenz.
FEBS Lett. 2004, 560, 141–146. [Google
Scholar] [CrossRef]
- Zheng, B. J.; Guan, Y.; Tang, Q.; Du, C.;
Xie, F. Y.; He, M. L.; Chan, K. W.; Wong, K.
L.; Lader, E.; Woodle, M. C.; et al.
Prophylaktische und therapeutische Wirkungen
kleiner störender RNA gegen
SARS-Coronavirus. Antivir. Res. 2004, 9,
365–374. [Google Scholar]
- Chang, Z.; Hu, J. Kann siRNA SARS erobern?
Gene Ther. 2006, 13, 871–872. [Google
Scholar] [CrossRef]
- Lu, C. Y.; Huang, H.-Y.; Yang, T. H.;
Chang, L. Y.; Lee, C. Y.; Huang, L. M.
siRNA-Stummschaltung des
Angiotensin-Converting-Enzyms 2 reduzierte
die mit dem akuten respiratorischen Syndrom
verbundenen Coronavirus-Replikationen in
Vero-Zellen. EUR. J. Clin. Microbiol.
Infizieren. Dis. 2008, 27, 709–715. [Google
Scholar] [CrossRef]
- Millet, J. K.; Nal, B. Untersuchung der
funktionellen Rolle von Wirtszellproteinen,
die an einer Coronavirus-Infektion beteiligt
sind, unter Verwendung eines
hochspezifischen und skalierbaren
RNA-Interferenz-Ansatzes (RNAi). Methoden
Mol. Biol. 2015, 1282, 231–240. [Google
Scholar] [PubMed]
- Nur, S. M.; Hasan, M. A.; Amin, M. A.;
Hossain, M.; Sharmin, T. Design potenzieller
RNAi (miRNA und siRNA) -Moleküle für das
Coronavirus (MERS-CoV) -Gen-Silencing des
respiratorischen Syndroms im Nahen Osten
mittels Computermethode. Interdiscip. Sci.
2015, 7, 257–265. [Google Scholar]
[CrossRef] [PubMed]
- McDonagh, P.; Sheehy, P. A.; Norris, J. M.
Eine kombinierte siRNA-Therapie gegen
Katzen-Coronavirus kann das Auftreten einer
antiviralen Resistenz in vitro verzögern.
Tierarzt. Microbiol. 2015, 176, 10–18.
[Google Scholar] [CrossRef] [PubMed]
- Gu, W. Y.; Li, Y.; Liu, B. J.; Wang, J.;
Yuan, G. F.; Chen, S. J.; Zuo, Y.Z.; Fan,
J.H. Kurze Haarnadel-RNAs, die auf M- und
N-Gene abzielen, reduzieren die Replikation
des Schweine-Deltacoronavirus in ST-Zellen.
Virus Genes 2019, 55, 795–801. [Google
Scholar] [CrossRef]
- Li, K.; Li, H.; Branche.; Song, D.; Zhang,
F.; Lei, D.; Luo, S.; Li, Z.; Gong, W.;
Huang, D.; et al. Signifikante Hemmung des
wieder aufgetauchten und neu auftretenden
enterischen Schweine-Coronavirus in vitro
unter Verwendung des multiplen
shRNA-Expressionsvektors. Virostatikum. Res.
2019, 166, 11–18. [Google Scholar]
[CrossRef]
- Sohrab, S. S.; El-Kafrawy, S. A.; Mirza,
Z.; Kamal, M. A.; Azhar, E.L. Design und
Lieferung von therapeutischen siRNAs:
Anwendung auf MERS-Coronavirus. Curr. Pharm.
Des. 2018, 24, 62–77. [Google Scholar]
[CrossRef]
- Delrue, I.; Verzele, D.; Madder, A.;
Nauwynck, H. J. Inaktivierte
Virusimpfstoffe: Von der Chemie zur
Prophylaxe: Verdienste, Risiken und
Herausforderungen. Expert Rev. Vaccines
2012, 11, 695–719. [Google Scholar]
[CrossRef]
- Deng, M. P.; Hu, Z. H.; Wang, H. L.; Deng,
F. Entwicklung von Untereinheiten- und
VLP-Impfstoffen gegen das Influenza-A-Virus.
Virol. Sünde. 2012, 27, 145–153. [Google
Scholar] [CrossRef]
- Decaro, N.; Campolo, M.; Mari, V.;
Desario, C.; Colaianni, M. L.; Di Trani, L.;
Cordioli, P.; Buonavoglia, C. Ein Kandidat
für einen modifizierten
Lebend-Coronavirus-Lebendimpfstoff:
Bewertung der Sicherheit und Immunogenität.
Neues Mikrobiol. 2009, 32, 109–132. [Google
Scholar]
- Bovilis® Coronavirus. Online verfügbar:
- https://www.merck-animal-health-usa.com/product/
- Rinder / Bovilis-Coronavirus / 1 (Zugriff
am 25. März 2020).
- Nemoto, M.; Kanno, T.; Bannai, H.;
Tsujimura, K.; Yamanaka, T.; Kokado, H.
Antikörperantwort auf Pferde-Coronavirus bei
Pferden, die mit einem
Rinder-Coronavirus-Impfstoff geimpft wurden.
Tierarzt. Med. Sci. 2017, 79, 1889–1891.
[Google Scholar] [CrossRef]
- Ibrahim, H. S.; Kafi, S.K. Ein Ansatz zur
rechnergestützten Impfstoffentwicklung für
MESR-CoV-Infektionen. Methoden Mol. Biol.
2020, 2131, 39–145. [Google Scholar]
[PubMed]
- Battacharaya, M.; Sharma, A. R.; Patra,
P.; Ghosh, P.; Sharma, G.; Patra, B. C.;
Lee, S. S.; Chakraborty, C. Entwicklung
eines Epitop-basierten Peptid-Impfstoffs
gegen das neuartige Coronavirus 2019
(SARS-CoV-2): Immunoinformatik-Ansatz. J.
Med. Virol. 2020. [Google Scholar]
[CrossRef] [PubMed]
- Tahir, U. I.; Qamar, M.; Saleem, S.;
Ashfaq, U. A.; Bari, A.; Anwar, F.;
Alqatani, S. Epitop-basiertes
Peptid-Impfstoffdesign und Darstellung der
Zielstelle gegen das Coronavirus des
respiratorischen Syndroms im Nahen Osten:
Eine Studie zur Immuninformatik. J. Transl.
Med. 2019, 17, 362. [Google Scholar]
[CrossRef]
- Srivastava, S.; Kamthania, M.; Singh, S.;
Saxena, A. K.; Sharma, N. Strukturelle
Grundlage für die Entwicklung eines
Multi-Epitop-Impfstoffs gegen das
Atemwegssyndrom im Nahen Osten unter
Verwendung des In-Silico-Ansatzes.
Infizieren. Arzneimittelresistenz. 2018, 11,
2377–2391. [Google Scholar] [CrossRef]
[PubMed]
- Ahmed, S. F.; Quadeer, A. A.; McKay, M. R.
Vorläufige Identifizierung potenzieller
Impfstoffziele für das COVID-19-Coronavirus
(SARS-CoV-2) basierend auf immunologischen
SARS-CoV-Studien. Viren 2020, 12, 254.
[Google Scholar] [CrossRef]
- Zakhartchouk, A. N.; Viswanathan, N.;
Moshynskyy, I.; Petric, M.; Babiuk, L. A.
Optimierung eines DNA-Impfstoffs gegen SARS.
DNA Cell Biol. 2007, 26, 721–726. [Google
Scholar] [CrossRef]
- Callendret, B.; Lorin, V.; Charneau, P.;
Marianneau, P.; Contamin, H.; Betton, J. M.;
van der Werf, S.; Escriou, N. Heterologe
virale RNA-Exportelemente verbessern die
Expression des Coronavirus-Spike-Proteins
des schweren akuten respiratorischen
Syndroms (SARS) und die Schutzwirkung von
DNA-Impfstoffen gegen SARS. Virology 2007,
363, 288–302. [Google Scholar] [CrossRef]
- Shim, B. S.; Park, S. M.; Quan, J. S.;
Jere, D.; Chu, H.; Song, M. K.; Kim, D. W.;
Jang, Y.S.; Yang, M. S.; Han, S. H.; et al.
Die intranasale Immunisierung mit
Plasmid-DNA, die für das Spike-Protein von
SARS-Coronavirus /
Polyethylenimin-Nanopartikeln kodiert, löst
Antigen-spezifische humorale und zelluläre
Immunantworten aus. BMC Immunol. 2010, 11,
65. [Google Scholar] [CrossRef]
- Chen, Y. N.; Wu, C. C.; Yeo, Y.; Xu, P.;
Lin, T.L. Eine
DNA-Prime-Protein-Boost-Impfstrategie, die
auf das
Puten-Coronavirus-Spike-Proteinfragment
abzielt, das neutralisierendes Epitop gegen
infektiöse Exposition enthält. Tierarzt.
Immunol. Immunopathol. 2013, 152, 359–369.
[Google Scholar] [CrossRef] [PubMed]
- Xue, R.; Tian, Y.; Zhang, Y.; Zhang, M.;
Tian, F.; Ma, J.; Jiang, S. Wirksamkeit
und Immunogenität eines lebenden L.
acidophilus, der das SAD-Epitop des
übertragbaren Gastroenteritis-Virus als
oralen Impfstoff exprimiert. Acta Virol.
2019, 63, 301–308. [Google Scholar]
[CrossRef] [PubMed]
- Nyon, M. P.; Du, L.; Tseng, C. K.; Seid,
C. A.; Pollet, J.; Naceanceno, K. S.;
Agrawal, A.; Algaissi, A.; Peng, B. H.; Tai,
W.; et al. Entwicklung einer stabilen
CHO-Zelllinie zur Expression eines
MERS-Coronavirus-Impfstoffantigens. Vaccine
2018, 36, 1853–1862. [Google Scholar]
[CrossRef] [PubMed]
- Li, H. Y.; Ramalingam, S.; Chye, M.L. Die
Akkumulation von rekombinantem
SARS-CoV-Spike-Protein in Cytosol und
Chloroplasten weist auf das Potenzial für
die Entwicklung pflanzlicher oraler
Impfstoffe hin. Exp. Biol. Med. 2006, 231,
1346–1352. [Google Scholar] [CrossRef]
- Pogrebnyak, N.; Golovkin, M.; Andrianov,
V.; Spitsin, S.; Smirnov, Y.; Egolf, R.;
Koprowski, H. Schwere Proteinproduktion des
akuten respiratorischen Syndroms (SARS) S in
Pflanzen: Entwicklung eines rekombinanten
Impfstoffs. Proc. Natl. Acad. Sci. USA 2005,
102, 9062–9067. [Google Scholar] [CrossRef]
- Zheng, N.; Xia, R.; Yang, C.; Yin, B.; Li,
Y.; Duan, C.; Liang, L.; Guo, H.; Xie, Q.
Verstärkte Expression des
SARS-CoV-Nucleocapsid-Proteins in Tabak und
dessen Immunogenität in Mäusen. Vaccine
2009, 27, 5001–5007. [Google Scholar]
[CrossRef]
- Do, V. T.; Jang, J.; Park, J.; Dao, H. T.;
Kim, K.; Hahn, T.W. Rekombinantes Adenovirus
mit einem kernneutralisierenden Epitop des
epidemischen Schweine-Durchfallvirus und dem
hitzelabilen Enterotoxin B von Escherichia
coli als Schleimhautimpfstoff. Bogen. Virol.
2020, 165, 609–618. [Google Scholar]
[CrossRef]
- Haagmans, B. L.; van den Brand, J. M.;
Raj, V. S.; Volz, A.; Wohlsein, P.; Smits,
S. L.; Schipper, D.; Bestebroer, T. M.;
Okba, N.; Fux, R.; et al. Ein Impfstoff auf
Orthopoxvirus-Basis reduziert die
Virusausscheidung nach einer
MERS-CoV-Infektion bei Dromedarkamelen.
Science 2016, 351, 77–81. [Google Scholar]
[CrossRef]
- Veit, S.; Jany, S.; Fux, R.; Sutter, G.;
Volz, A. CD8 + T-Zellen, die auf das
Coronavirus-Nucleocapsid des
respiratorischen Syndroms im Nahen Osten
reagieren, das von Vaccinia-Virus-MVA in
Mäusen abgegeben wird. Viren 2018, 10, 718.
[Google Scholar] [CrossRef]
- Faber, M.; Lamirande, E. W.; Roberts, A.;
Rice, A. B.; Koprowski, H.; Dietschold, B.;
Schnell, M. J. Eine einzelne Imunisierung
mit einem Vektor auf Rhabdovirus-Basis, der
das Coronavirus (SARS-CoV) S-Protein des
schweren akuten respiratorischen Syndroms
exprimiert, führt zur Produktion hoher
Mengen an SARS-CoV-neutralisierenden
Antikörpern. J. Gen. Virol. 2005, 86,
1435–1440. [Google Scholar] [CrossRef]
- Deming, D.; Sheahan, T.; Heise, M.; Yount,
B.; Davis, N.; Sims, A.; Suthar, M.;
Harkema, J.; Whitmore, A.; Pickles, R.; et
al. Impfstoffwirksamkeit bei seneszenten
Mäusen, die mit rekombinanten
SARS-CoV-infizierten epidemischen und
zoonotischen Spike-Varianten in Kontakt
gebracht wurden. PLoS Med. 2006, 3, e525.
[Google Scholar] [CrossRef] [PubMed]
- Sheahan, T.; Whitmore, A.; Long, K.;
Ferris, M.; Rockx, B.; Funkhouser, B.;
Donaldson, E.; Gralinski, L.; Collier, M.;
Heise, M.; et al. Erfolgreiche
Impfstrategien, die gealterte Mäuse vor
tödlichen Herausforderungen durch
Influenzaviren und heterologes Coronavirus
mit schwerem akutem respiratorischem Syndrom
schützen. J. Virol. 2011, 85, 217–230.
[Google Scholar] [CrossRef] [PubMed]
- Bennett, R. P.; Dalby, B. Proteinabgabe
unter Verwendung von VP22. Nat. Biotechnol.
2002, 20, 20. [Google Scholar] [CrossRef]
[PubMed]
- Almazan, F.; Galan, C.; Enjuanes, L.
Engineering infektiöser cDNAs des
Coronavirus als bakterielle künstliche
Chromosomen. Methoden Mol. Biol. 2008, 454,
275–291. [Google Scholar] [PubMed]
- Shanmugarai, B.; Malla, A.; Phoolcharoen,
W. Entstehung des neuartigen Coronavirus
2019-nCoV: Notwendigkeit einer schnellen
Entwicklung von Impfstoffen und Biologika.
Pathogens 2020, 9, 148. [Google Scholar]
[CrossRef] [PubMed]
- Lundstrom, K. RNA-Viren als Werkzeuge in
der Gentherapie und Impfstoffentwicklung.
Gene 2019, 10, 189. [Google Scholar]
[CrossRef]
- Lundstrom, K. Plasmid-DNA-basierte
Alphavirus-Impfstoffe. Impfstoffe 2019, 7,
29. [Google Scholar] [CrossRef]
- Francesco, N. Spielt die
antikörperabhängige Verstärkung eine Rolle
bei der COVID-19-Pathogenese? Schweizerisch.
Med. Wkly. 2020, 150, w20249. [Google
Scholar]
- Tetro, J.A. Empfängt COVID-19 ADE von
anderen Coronaviren? Mikroben. Inf. 2020,
22, 72–73. [Google Scholar] [CrossRef]
- Wan, Y.; Shang, J.; Sun, S.; Tan, W.;
Chen, J.; Geng, Q.; He, L.; Chen, Y.; Wu,
J.; Shi, Z.; et al. Molekularer Mechanismus
zur antikörperabhängigen Verbesserung des
Coronavirus-Eintritts. J. Virol. 2020, 94,
e02015-19. [Google Scholar] [CrossRef]
- Chen, K.; Kolls, J.K. T-Zell-vermittelte
Immunabwehr des Wirts in der Lunge. Annu.
Rev. Immunol. 2013, 31, 605–633. [Google
Scholar] [CrossRef] [PubMed]
- Mason, R.J. Pathogenese von COVID-19 aus
zellbiologischer Sicht. EUR. Respir. J.
2020, 55, 2000607. [Google Scholar]
[CrossRef] [PubMed]
- Bloch, E. M.; Shoham, S.; Casadevall, A.;
Sachais, B. S.; Shaz, B.; Winters, J. L.;
van Burskirk, C.; Grossman, B. J.; Joyner,
M.; Henderson, J. P.; et al. Einsatz von
Rekonvaleszenzplasma zur Vorbeugung und
Behandlung von COVID-19. J. Clin. Investig.
2020, 138745. [Google Scholar] [CrossRef]
[PubMed]
- Zhang, L.; Pang, R.; Xue, X.; Bao, J.;
Ja.; Daj, Y.; Zheng, Y.; Fu, Q.; Hu, Z.; Yi,
Y. Anti-SARS-CoV-2-Virus-Antikörperspiegel
in Rekonvaleszenzplasma von sechs Spendern,
die sich von COVID-19 erholt haben. Altern
2020, 12. [Google Scholar] [CrossRef]
- Shen, C.; Wang, Z.; Zhao, F.; Yang, Y.;
Li, J.; Yuan, J.; Wang, F.; Li, D.; Yang,
M.; Xing, L.; et al. Behandlung von 5
kritisch kranken Patienten mit COVID-19 mit
Rekonvaleszenzplasma. JAMA 2020. [Google
Scholar] [CrossRef]
- Grein, J.; Ohmagari, N.; Shin, D.; Diaz,
G.; Asperges, E.; Castagna, A.; Feldt, T.;
Green, G.; Green, M. L.; Lescure, F. X.; et
al. Mitfühlende Anwendung von Remdesivir bei
Patienten mit COVID-19. N. Engl. J. Med.
2020. [Google Scholar] [CrossRef]
- Online verfügbar:
www.gilead.com/news-and-press/company-statements/gilead-sciences-statement-on-data-from-remdesivir-study-in-patients-with-
schwere Covid-19-in-China (Zugriff am 30.
April 2020).
- Online verfügbar:
www.theguardian.com/world/2020/apr/23/high-hopes-drug-for-covid-19-treatment-failed-in-full-trial
(Zugriff am 30. April 2020).
- Ferner, R. E.; Aronson, J.K. Chloroquin
und Hydroxychloroquin in COVID-19. BMJ 2020,
369, m1432. [Google Scholar] [CrossRef]
- Gao, J.; Tian, Z.; Yang, X. Durchbruch:
Chloroquinphosphat hat in klinischen Studien
eine offensichtliche Wirksamkeit bei der
Behandlung von COVID-19-assoziierter
Pneumonie gezeigt. Biosci. Trends 2020, 14,
72–73. [Google Scholar] [CrossRef]
- Chen, Z.; Hu, J.; Zhang, Z.; Jiang, S.;
Han, S.; Yan, D.; Zhuang, R.; Hu, B.; Zhang,
Z. Wirksamkeit von Hydroxychloroquin bei
Patienten mit COVID-19: Ergebnisse einer
randomisierten klinischen Studie. Version 2.
medRxiv 2020. [Google Scholar] [CrossRef]
- Yan, D.; Zhang, Z. Therapeutische Wirkung
von Hydroxychloroquin auf neuartige
Coronavirus-Pneumonie (COVID-19).
Chinesisches Register für klinische Studien.
Online verfügbar: http://www.chictr.org.cn/
- showproj.aspx? proj = 48880 (Zugriff am
28. April 2020).
- Murphy, M.; Carmichael, A.J. Tödliche
toxische epidermale Nekrolyse in Verbindung
mit Hydroxychloroquin. Clin. Exp. Dermatol.
2001, 26, 457–458. [Google Scholar]
[CrossRef]
- Makin, A. J.; Wendon, J.; Fitt, S.;
Portmann, B. C.; Williams, R. Fulminantes
Leberversagen infolge von Hydroxychloroquin.
Gut 1994, 35, 569–570. [Google Scholar]
[CrossRef] [PubMed]
- Chorin, E.; Dai, M.;
Shulman, E.; Wadhwani, L.; Bar-Cohen, R.;
Barbhayia, C.; Aizer, A.; Holmes, D.;
Bernstein, S.; Spinelli, M.; et al. Das
QT-Intervall bei Patienten mit
SARS-CoV-2-Infektion, die mit
Hydroxychloroquin / Azithromycin behandelt
wurden. medRxiv 2020. [Google Scholar]
[CrossRef]
- Li, Y.; Xie, Z.; Lin, W.; Cai, W.; Wen,
C.; Guan, Y.; Mo, X.; Wang, J.; Wang, Y.;
Peng, P.; et al. Eine explorative
randomisierte, kontrollierte Studie zur
Wirksamkeit und Sicherheit von Lopinavir /
Ritonavir oder Arbidol bei erwachsenen
Patienten, die mit leichtem / mittelschwerem
COVID-19 (ELACOI) ins Krankenhaus
eingeliefert wurden. medRxiv 2020. [Google
Scholar] [CrossRef]
- Online verfügbar: www.who.int/blueprint/
- Prioritätskrankheiten / Schlüsselaktion /
Roman-
- coronavirus-Landschaft-ncov.pdf (Zugriff
am 28. April 2020).
- Zhao, H. COVID-19 treibt Fledermäuse in
China vor neue Bedrohungen. Science 2020,
367, 1436. [Google Scholar]
- Cyranoski, D. Das Geheimnis über die
tierische Quelle des Coronavirus vertieft
sich. Nature 2020, 579, 18–19. [Google
Scholar] [CrossRef]
© 2020 vom Autor. Lizenznehmer MDPI, Basel,
Schweiz. Dieser Artikel ist ein
Open-Access-Artikel, der unter den Bedingungen
der CC BY-Lizenz (Creative Commons
Attribution)
(http://creativecommons.org/licenses/by/4.0/)
verbreitet wird.
|

 https://www.youtube.com/watch?v=FowK14stnlk
https://www.youtube.com/watch?v=FowK14stnlk https://www.youtube.com/watch?v=Trl2vrqNkNM
https://www.youtube.com/watch?v=Trl2vrqNkNM